Cell Research|高绍荣\张勇\高亚威合作阐明小鼠受精过程中核小体排布建立模式
真核生物的染色体通常呈现高度凝集的状态,而核小体是染色体结构中最基本的重复单元,由组蛋白八聚体缠绕着约147bp DNA组成。核小体具有致密的结构,阻止了一些特异性DNA序列识别蛋白的结合,因此核小体的排布与染色体可及性、基因转录活性密切相关[1]。对于包括人类、小鼠在内的哺乳动物,精子的发生过程会经历组蛋白被鱼精蛋白替换的独特事件,这促使精子基因组进一步的压缩、凝集,而当受精作用发生后,鱼精蛋白迅速解聚,组蛋白与基因组DNA重新组装为核小体结构,这种染色质重塑对于合子基因组激活(zygotic genome activation, ZGA)等转录事件有着重要调控作用[2],然而受精后核小体重建、重排的偏好模式和影响机制尚不明晰。
同济大学高绍荣教授及张勇教授课题组近年来有多项合作工作致力于探究小鼠早期胚胎发育过程中的表观遗传调控,尤其是ZGA和谱系分化相关的转录调控,例如2016年发表于Nature杂志的H3K4me3、H3K27me3修饰建立与小鼠胚胎基因转录调控相关研究[3],以及2018年发表于Nature Cell Biology杂志的H3K9me3参与逆转座子沉默和谱系分化相关研究[4]。这些工作均利用极低起始量的染色质免疫共沉淀-测序(Ultra-low-input micrococcal nuclease-based native ChIP-seq, ULI-NChIP-seq)技术绘制了重要组蛋白甲基化修饰在早期胚胎发育中的动态变化图谱,也进一步强调了以组蛋白为基础的染色质状态调控对于正常发育过程的重要意义。
4月15日,高绍荣教授和张勇教授课题组合作,在Cell Research杂志在线发表题为“Dynamic nucleosome organization after fertilization reveals regulatory factors for mouse zygotic genome activation”的文章。利用低起始量的微球菌核酸酶高通量测序(ULI-MNase-seq),首次检测了小鼠受精后12h内雌雄原核连续时间点的核小体排布模式建立过程,探讨了影响核小体排布的潜在机制,并基于核小体建立模式的动态变化鉴定了影响合子基因组激活的潜在转录因子。

在该研究中,研究人员通过显微操作,分离获得了小鼠雌雄配子,以及受精后0.5小时至12小时9个连续时间点的雌雄原核,并对配子和雌雄原核的核小体排布情况,使用ULI-MNase-seq技术进行了全基因组的检测(图1a)。研究人员同时开发了一套针对核小体数据的分析流程NEPTUNE,基于此流程,研究人员分析发现:受精后雄原核基因组上鱼精蛋白迅速替换为核小体,在1h左右基本完成替换,而雌原核及使用圆形精子受精得到的雌雄原核不存在这种现象(图1b);在核小体占有率建立完毕之后,雌雄原核的核小体定位信号均发生了逐步重塑,核小体缺失区域(Nucleosome depleted region,NDR)在启动子、增强子开始建立,雄原核的NDR建立比雌原核要更为迅速,并且ZGA相关的基因相比于其他基因的NDR建立更为明显。上述分析说明,核小体排布的建立具有雌雄差异,并很可能反应了早期雌雄原核基因转录活性的区别。
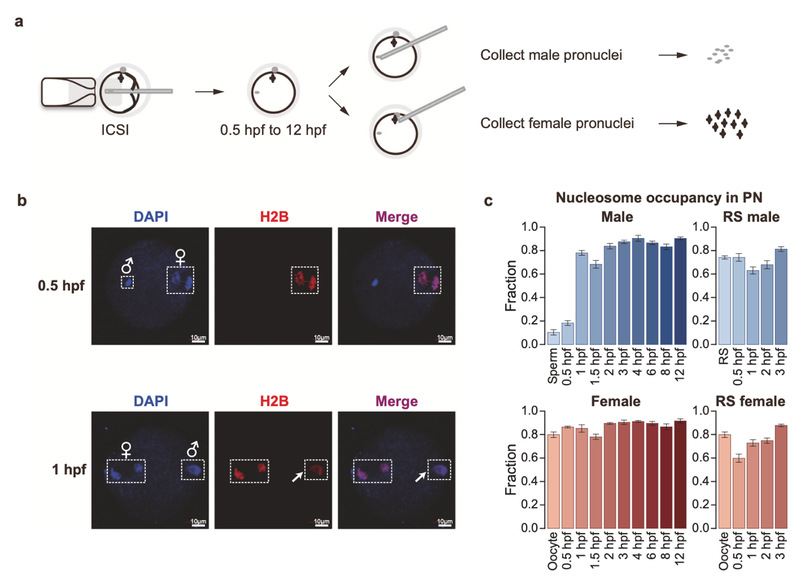
图1 小鼠雌雄原核受精过程中核小体排布模式迅速建立
研究人员进一步探索了影响早期核小体排布的因素,分析发现,GC含量与核小体占有(Nucleosome occupancy)的建立密切相关,GC含量高的区域倾向于在更早的时期建立核小体。进一步,研究人员发现抑制DNA复制及转录并不能对核小体占有率及排布模式产生显著影响,而使用HDAC抑制组蛋白乙酰化修饰可以明显降低+1核小体及NDR的形成,暗示乙酰化修饰介导的早期染色质重塑复合物的结合可能对核小体排布的建立起到了重要调控作用。
转录因子通常需要结合在核小体缺失区域发挥作用,因此分析转录因子结合区域核小体排布的变化,极有可能反应转录因子在基因组上的结合情况。研究人员基于核小体排布信号,计算了122个早期表达的转录因子模体上NDR的变化情况,发现其中一类转录因子的NDR在受精过程中发生了从无到有的建立(图2,Cluster 3),暗示这些因子极有可能在早期结合到基因组,并可能参与了合子基因组的激活。研究人员敲降了其中两个转录因子Mlx及Rfx1,发现Mlx可以调控合子时期ZGA的发生,而Rfx1可以显著影响2-细胞时期的ZGA。
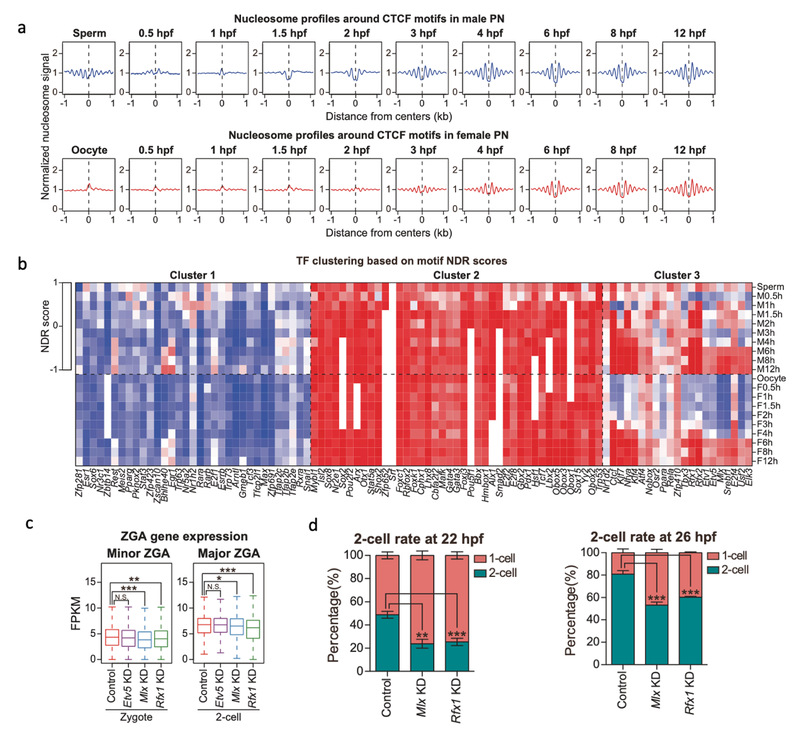
图2 基于雌雄原核核小体定位动态变化鉴定Mlx及Rfx1为ZGA关键因子
综上所述,该研究首次绘制了小鼠雌雄原核受精过程中核小体排布高分辨率图谱,发现了核小体排布的建立模式规律,并基于核小体排布模式的动态变化,鉴定了合子基因组激活过程中的关键转录因子。对于人们理解表观遗传学修饰在代际之间的传递,及对胚胎发育过程中基因表达的调控作用提供了有力的支持。
同济大学王晨飞研究员、高绍荣教授实验室的陈川博士、刘晓雨教授、李翀副研究员为本文的共同第一作者。高绍荣教授、张勇教授及高亚威教授为本文的共同通讯作者。该研究得到了科技部、基金委和上海市科委项目支持。
1.Struhl K, Segal E. Determinants of nucleosome positioning. Nat Struct Mol Biol 2013; 20:267-273.
2.Okada Y, Yamaguchi K. Epigenetic modifications and reprogramming in paternal pronucleus: sperm, preimplantation embryo, and beyond. Cell Mol Life Sci 2017; 74:1957-1967.
3.Liu X, Wang C, Liu W, Li J, Li C, Kou X, Chen J, Zhao Y, Gao H, Wang H, Zhang Y, Gao Y, et al. Distinct features of H3K4me3 and H3K27me3 chromatin domains in pre-implantation embryos. Nature 2016; 537:558-562.
4.Wang C, Liu X, Gao Y, Yang L, Li C, Liu W, Chen C, Kou X, Zhao Y, Chen J, Wang Y, Le R, et al. Reprogramming of H3K9me3-dependent heterochromatin during mammalian embryo development. Nat Cell Biol 2018; 20:620-631.
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


