Cell Reports Methods|王晨飞团队合作开发基于人工智能和大数据整合的单细胞注释工具SELINA
单细胞RNA测序技术的迅速发展为揭示复杂组织内的异质性提供帮助。在scRNA-seq数据处理中,细胞类型注释是解释功能特征和下游分析的关键步骤。细胞类型注释主要有两种策略:基于标记基因和基于参考数据集。基于参考数据集的策略使用机器学习方法将细胞类型标签从参考数据集转移到查询数据集,具有无偏、定量的优点,随着scRNA-seq数据量的积累,获得了更高的准确性和广泛的应用。然而,这一策略仍然存在诸多挑战,包括公共数据注释不统一、细胞数量不平衡、批次效应以及对参考数据集质量、数量的依赖。解决这些挑战对于提高细胞类型注释的准确性并充分发挥scRNA-seq数据的潜力至关重要。
近日,同济大学王晨飞、张敬课题组和四川大学华西口腔医院李太文课题组合作在Cell Reports Methods杂志上发表文章Single-cell Assignment using Multiple-Adversarial Domain Adaptation Network with Large-scale References,开发了基于AI和大数据整合的单细胞注释工具SELINA。

SELINA首先收集处理和注释了公共数据库中海量的人类组织单细胞数据,提供全面、统一的基因表达参考图谱,包含170万个细胞,涵盖230种人类细胞类型。在此基础上,SELINA基于AI技术对数据进行预训练,基于多对抗域适应网络(Multiple-Adversarial Domain Adaptation, MADA)2消除参考数据集中的批次效应,并通过合成少数过采样技术(SMOTE)1优化少数细胞类型的注释,最后,SELINA使用自动编码器将查询数据与参考数据进行拟合,去除查询数据和参考数据集的批次效应。基于预训练的模型,SELINA可快速有效的将参考数据集细胞注释标签转移到查询数据集,实现细胞类型注释(图1)。研究团队在大多数人体组织中证明了它的稳健性和优越性,并且可以准确注释各种疾病场景下的细胞。SELINA提供了python/R软件包,以及在线注释分析网站(http://selina.compbio.cn/#/Annotation),为人类单细胞RNA-seq数据注释提供了完整的解决方案。
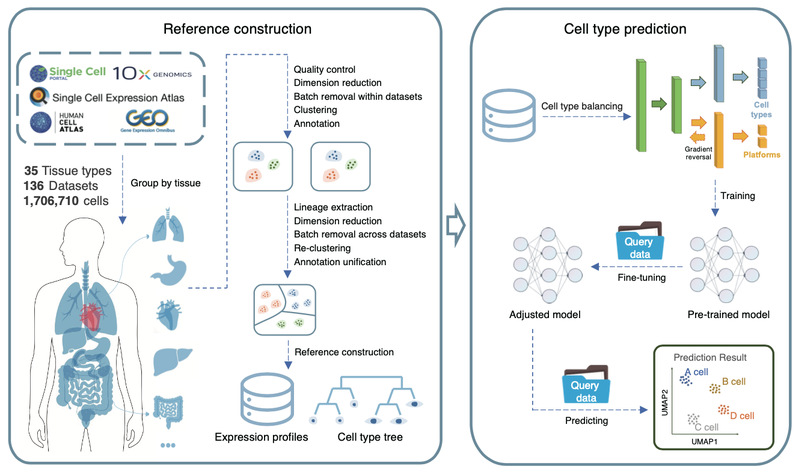
图 1 SELINA的数据集构建及算法结构
(图源:Ren P & Shi X, et al., Cell Reports Methods 2023)
SELINA具有优秀的批次效应去除效果
SELINA的预训练框架的架构由三个组件组成:特征提取器、细胞类型鉴别器和测序平台鉴别器。理想情况下,如果去除了测序平台不同造成的批次效应,不同平台测序的共有细胞类型会聚集得更近,而特异细胞类型会进一步分离。研究人员在五个不同的肝脏数据集上分别计算了不同测序平台共有细胞和平台特异细胞的局部逆辛普森指数(LISI)得分3(较高的LISI得分意味着细胞在不同平台上可以很好地混合)。特征提取器转换和细胞类型分类器转换都提高了共有细胞的LISI得分,并降低了平台特定细胞的LISI得分(图2a)。与原始嵌入相比,来自不同平台的相同细胞类型细胞聚集得更近,这表明利用细胞类型特异的平台鉴别器,特征提取器可以揭示每个细胞类型内部潜在共同特征。SELINA的预训练框架显著消除了
批次效应,并且与传统批次校正工具Harmony相比,在CD4+T细胞、CD8+T细胞和NK细胞等类似细胞类型中显示出更好的分离效果(图2b-d)。
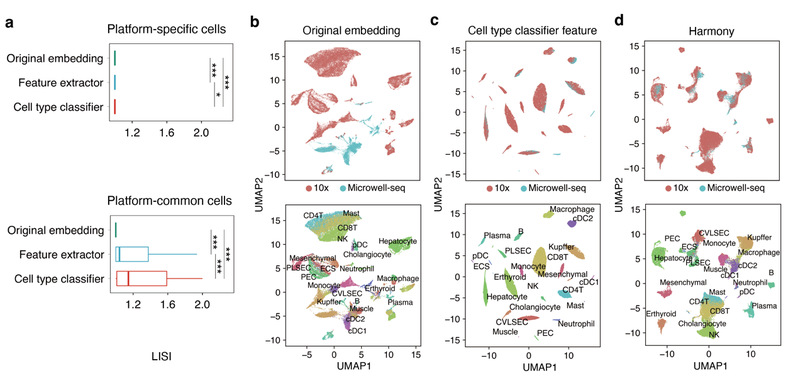
图 2基于原始数据和利用SELINA及Harmony去除批次效应之后的细胞分布
(图源:Ren P & Shi X, et al., Cell Reports Methods 2023)
SELINA在综合性能评估中优于其他现有工具
SELINA分别使用来自单个测序平台和多个测序平台的数据,系统地将SELINA与现有注释工具和传统机器学习方法的性能进行比较。单平台评估对来自9个组织的使用相同测序平台的多数据集进行了测试,数据覆盖479,740个细胞。多平台评估对来自14个组织的使用不同测序平台的多数据集进行了测试,数据覆盖658,270个细胞。对于每个组织,挑选一个数据集作为查询数据,而其他数据集被合并作为参考数据。重复训练和测试过程,直到测试每个数据集。这一个组织中所有数据集的平均准确度和MacroF1代表了预测性能。SELINA达到最佳准确度MacroF1在单平台和多平台数据上的评估。对于单平台测试(图3a),SELINA是以平均准确率为61.51%排名第一。在多平台评估方面(图3b),SELINA以平均准确率64.42%排名第一。这些结果表明SELINA在细胞注释任务上具有鲁棒性和优越性。
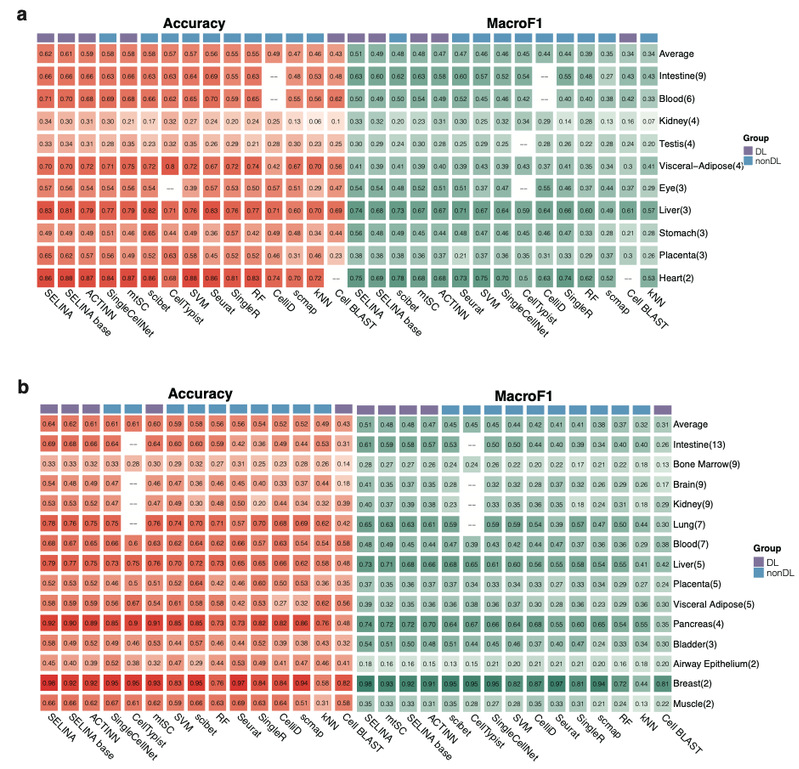
图 3 SELINA在综合性能评估中优于其他现有工具
(图源:Ren P & Shi X, et al., Cell Reports Methods 2023)
SELINA可以使用自定义数据集进行注释
为探究SELINA在用户自定义数据集的表现,研究人员在Allen Brain Atlas4,5的2个数据集上测试了各个工具的预测情况,SELINA在9个测试工具中排名第二,平均准确率为93.32%,平均MacroF1得分为0.9336(图4a)。高预测精度表明SELINA在用更精细注释的细胞类型数据集进行注释时候方面具有优异表现(图4b,c)。综上所述,SELINA可以使用来自具有专家知识的用户自定义数据集作为参考进行细胞注释。
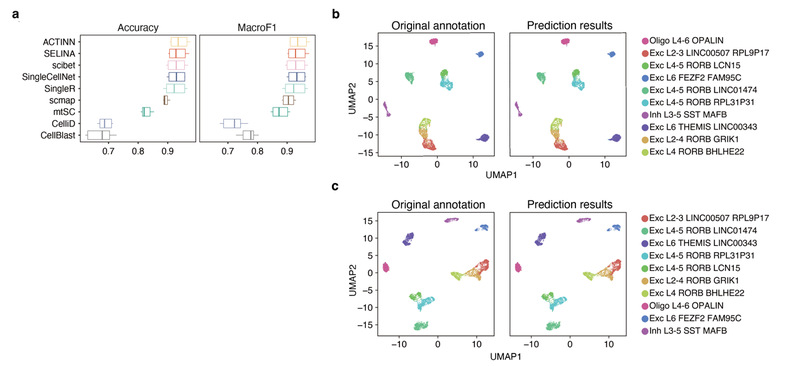
图4 SELINA在用户自定义数据中表现优秀
(图源:Ren P & Shi X, et al., Cell Reports Methods 2023)
SELINA可以对疾病数据进行注释
为探究SELINA是否可以用于注释不同疾病场景下的细胞,研究人员使用正常免疫和组织特异性细胞构建了参考数据集,并将SELINA与其他注释工具在II型糖尿病(T2D)、非小细胞肺癌(NSCLC)和阿尔茨海默病(AD)等疾病场景中的性能进行了比较。SELINA在T2D数据集、NSCLC数据集和AD数据集上均表现出了高于其他工具的性能(图5a-f)。疾病相关细胞中基因表达的改变可能与其疾病阶段相关。为了研究SELINA是否可以追踪疾病相关细胞中的表达差异,我们选择了来自T2D数据的β细胞、来自NSCLC的恶性细胞和来自AD的神经元细胞来评估,因为它们是相应疾病中的主要异常细胞。在T2D数据集中,当HbA1c值(衡量患2型糖尿病风险的指标)上升时,准确预测的β细胞比例进一步下降(图5g)。在NSCLC数据集中,准确预测的细胞百分比在疾病进展过程中下降(图5h)。在AD数据集中,准确预测的神经元的百分比也随着braak阶段而下降(图5i)。上述分析表明SELINA对疾病阶段敏感,能够准确区分正常细胞和患病细胞之间的差异。
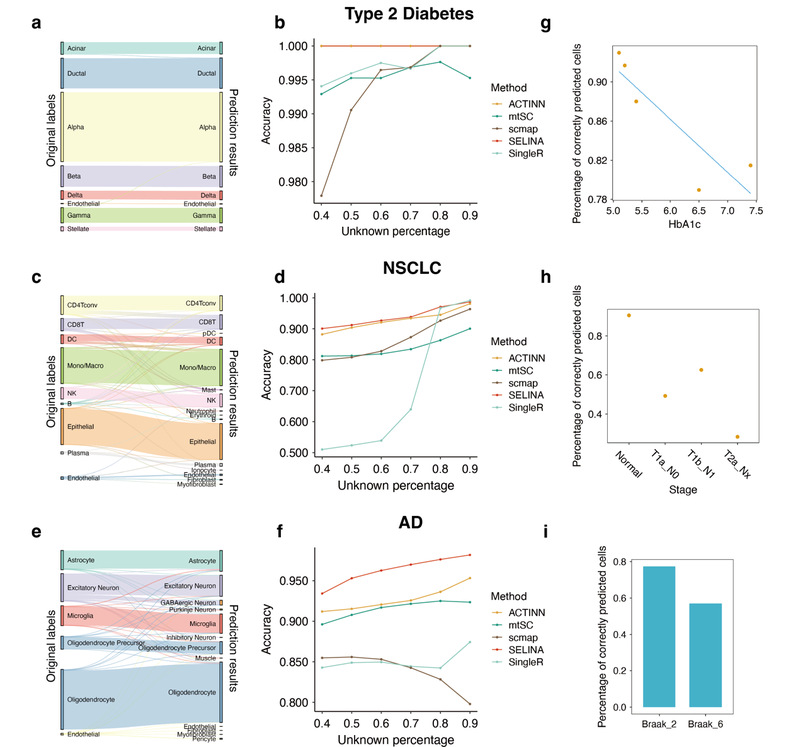
图 5 SELINA可用于疾病数据注释
(图源:Ren P & Shi X, et al., Cell Reports Methods 2023)
总的来说,SELINA通过人工智能算法整合海量单细胞数据,在单细胞注释这一任务上取得了优于现有算法的效果。然而这一框架目前也存在一定局限性,如MADA网络提取的数据特征不具有可解释性,注释效果受初始数据集注释质量影响等。未来团队将致力于开发基于海量数据整合及具备可解释性的人工智能算法,在高效注释细胞类型的同时挖掘细胞类型的形成机制。
同济大学生命科学与技术学院王晨飞教授、张敬教授,四川大学华西口腔医院李太文副研究员为该论文的共同通讯作者,同济大学研究生任鹏飞、石笑颖为共同第一作者。该工作得到了国家科技部重点研发、国家自然科学基金委、教育部细胞干性与命运编辑前沿中心、上海市自主智能无人系统科学中心等项目的重要支持。
参考文献
1.Chawla, N.V., Bowyer, K.W., Hall, L.O., and Kegelmeyer, W.P. (2002). SMOTE: Synthetic Minority Over-sampling Technique. J Artif Intell Res 16, 321-357.
2.Ren, P., Shi, X., Dong, X., Yu, Z., Ding, X., Wang, J., Sun, L., Yan, Y., Hu, J., Zhang, P., et al. (2022). SELINA: Single-cell Assignment using Multiple-Adversarial Domain Adaptation Network with Large-scale References. bioRxiv, 2022.2001.2014.476306. 10.1101/2022.01.14.476306.
3.Frumkin, D., Wasserstrom, A., Itzkovitz, S., Harmelin, A., Rechavi, G., and Shapiro, E. (2008). Amplification of multiple genomic loci from single cells isolated by laser micro-dissection of tissues. BMC Biotechnol 8, 17. 10.1186/1472-6750-8-17.
4.Tasic, B., Yao, Z., Graybuck, L.T., Smith, K.A., Nguyen, T.N., Bertagnolli, D., Goldy, J., Garren, E., Economo, M.N., and Viswanathan, S. (2018). Shared and distinct transcriptomic cell types across neocortical areas. Nature 563, 72-78.
5.Bakken, T.E., Jorstad, N.L., Hu, Q., Lake, B.B., Tian, W., Kalmbach, B.E., Crow, M., Hodge, R.D., Krienen, F.M., and Sorensen, S.A. (2021). Comparative cellular analysis of motor cortex in human, marmoset and mouse. Nature 598, 111-119.
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


