Science Advances | 刘琦教授团队开发基于人工智能度量学习的单细胞类型鉴定新方法
单细胞测序技术有助于刻画细胞层面的异质性,为进一步探索细胞功能和内在作用机制等奠定了基础。在单细胞转录组测序数据的分析过程中,细胞类型鉴定是必须且重要的一步。常规的细胞类型鉴定依赖于细胞类型标记基因,这种方法费时费力且不稳定,可重复性差,易导致不同实验室的鉴定结果不可比较。随着单细胞测序技术的迅猛发展,众多科学家联名在2017年提出人类细胞图谱计划(The Human Cell Atlas[1]),于2020年提出了人类癌症图谱计划(The Human Tumor Atlas[2]),海量的已标注细胞类型的单细胞转录组测序数据越来越多,利用这些数据作为参考数据集(reference)来鉴定新测序的细胞(single cell assignment against the reference),正成为一种细胞注释的新思路。
日前,同济大学生命科学与技术学院生物信息系刘琦教授课题组于10月30日在Science子刊Science Advances线上发表了题为“Learning for single cell assignment”的论文[3],发布了基于人工智能度量学习的单细胞类型鉴定新方法scLearn,以及简单易用的R包平台和包含人类、小鼠在内的多个组织器官的预训练的完整reference数据集,为有效利用海量的单细胞测序数据进行细胞类型鉴定提供了可借鉴的工具和资源。作者同时首次提出了一种基于最大化依赖的多标签维度约简策略(MDDM,multi-label dimension reduction via dependence maximization),用以针对多标签细胞类型鉴定进行度量学习,可适用于附加时间维度信息的单细胞类型鉴定。

利用参考数据集来鉴定新的细胞类型的方法具有两个方面的需求:(1)如果query细胞的细胞类型存在于reference中,那么需要以尽可能高的准确率对这些query细胞匹配正确的细胞类型(single cell assignment in the positive control scenario);(2)如果query细胞的细胞类型在reference中缺失,也需要正确的以“unassigned”的标识识别出来(single cell assignment in the negative control scenario)。但是现有的方法普遍存在两个不足,一是对于以上两个方面的要求往往不能同时兼顾;二是鲁棒性不佳,无法适用于多个来源的数据集。究其原因,作者认为现有的方法在框架设计上存在两方面的共性问题,一是衡量相似性的测度是人为选定的或者固定的,二是判定“unassigned”的阈值也是人为选定的。单细胞转录组测序数据具有高异质性和高噪音等特点,需要避免人为设定相应的测度和参数,而通过数据本身进行度量学习,将有助于解决该类共性问题。
基于以上考量,作者提出了一种基于人工智能度量学习的细胞类型鉴定框架scLearn,该框架将可以适用于单标签以及多标签的细胞类型鉴定。其核心思想是学习一个优化的转化矩阵,将reference数据集映射到特定的特征子空间中,在这个子空间中,相同类型细胞之间距离更近,不同类型细胞之间距离更远,使得转化后的reference数据更有利于进行细胞间相似性的准确衡量(图1)。
该工作通过在多达30套的单细胞benchmark数据集上的基准测试,证明了基于度量学习的scLearn具有普适而优良的细胞类型鉴定性能。scLearn计算平台基于R语言开发,同时提供了多达30套预训练的高质量的单细胞转录组测序reference数据集,其中包含了小鼠和人类的脑细胞,胰腺细胞,免疫细胞等等各个组织以及小鼠20个器官的数据,方便用户后续进行相关领域的研究。需要特别指出的是,该工作所提出的针对于附加时序信息的单细胞测序数据的细胞类型鉴定的新策略具有广阔的应用前景。以胚胎发育为例,除了需要鉴定细胞类型之外,鉴定其所处的发育阶段也是至关重要的问题。一个细胞的类型往往与其所处的发育阶段息息相关,scLearn可以充分利用这两类标签(发育阶段和细胞类型)之间的关系,同时进行两类标签的鉴定,提高了多维度下细胞注释的性能。
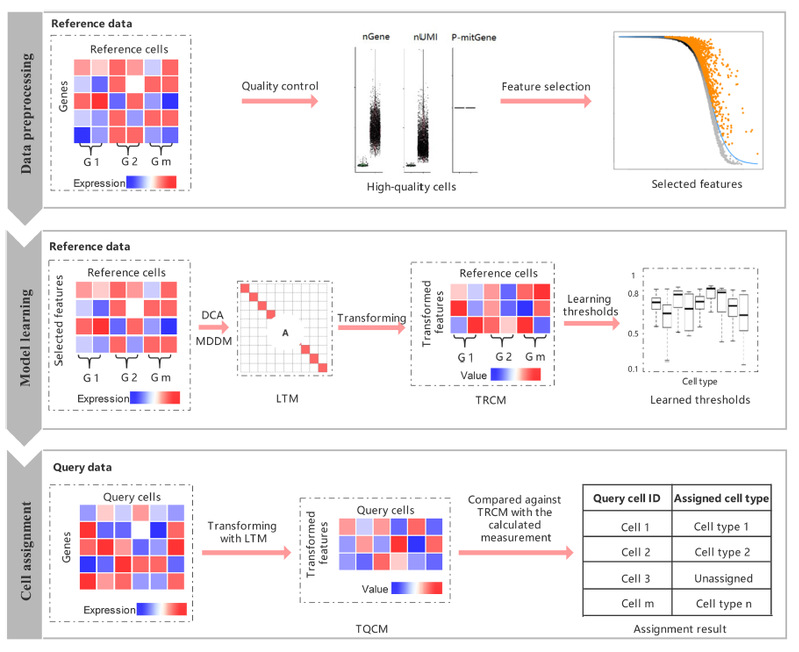
图1. scLearn的算法框架
刘琦教授课题组长期从事基于人工智能和组学数据分析的肿瘤精准治疗、药物发现以及基因编辑方面的研究工作。近年来关注的研究方向之一是基于人工智能面向单细胞测序数据开发组学分析平台及其在肿瘤免疫和细胞治疗领域的应用。2019年,刘琦教授课题组基于人工智能主题学习(Topic model)模型,在Nature Communications发表了针对于单细胞CRISPR筛选数据进行分析的计算平台MUSIC (Model-based Understanding of single cell CRISPR screening)[4]。本次工作是刘琦教授课题组继2019年MUSIC工作之后在单细胞测序数据分析领域的又一有益探索。该论文第一作者是刘琦教授课题组的博士生段斌,通讯作者是刘琦教授。该研究成果得到国家科技部精准医学重点研发计划,国家科技部慢病项目重点研发计划、国家自然科学基金面上项目以及上海市人工智能技术标准专项项目的资助。
1.Regev, A., et al., The Human Cell Atlas. Elife, 2017. 6.
2.Rozenblatt-Rosen, O., et al., The Human Tumor Atlas Network: Charting Tumor Transitions across Space and Time at Single-Cell Resolution. Cell, 2020. 181(2): p. 236-249.
3.Duan, B., et al., Learning for single cell assignment. Science Advance, 2020.
4.Duan, B., et al., Model-based understanding of single-cell CRISPR screening. Nat Commun, 2019. 10(1): p. 2233.
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


