Genome Biology | 王晨飞课题组开发单细胞分辨率的跨模态生成算法Cisformer解析转录调控机制
单细胞多组学技术能在同一细胞内测量多种模态,例如SHARE-seq方法可同时获取基因表达与染色质可及性信息1。虽然单细胞多组学方法能帮助我们理解细胞不同层面的相互调控机制,但是它面临实验复杂性高、成本昂贵以及数据信噪比低等诸多挑战。因此,从生物信息学的角度出发,通过构建计算模型,利用已知模态的信息生成未知模态数据,是一种有效可行的解决方法。目前,已有算法可用于单细胞数据的跨模态生成任务,比如BABEL2、Polarbear3和scButterfly4等,但是模型的泛化能力与可解释性仍有待加强。此外,如何通过模型帮助理解转录调控的复杂机制也是一个非常重要的问题。

2025年10月6日,同济大学生命科学与技术学院王晨飞团队在Genome Biology上发表了文章Cisformer: a scalable cross-modality generation framework for decoding transcriptional regulation at single-cell resolution。该研究开发了基于跨注意力机制的生成式人工智能模型Cisformer(https://github.com/wanglabtongji/Cisformer),用于单细胞分辨率的基因表达与染色质可及性数据的生成。综合考虑模型的复杂性与可解释性,Cisformer采用“decoder-only”的模型架构,创新性地提出了对染色质可及性数据相关的超长序列使用特征重复与选择以及新型索引编码处理。Cisformer具有优秀的单细胞数据生成能力与模型可解释性,能在癌症发生与器官衰老等重要生物学过程中鉴定潜在细胞类型特异的重要调控元件与转录因子(图1)。
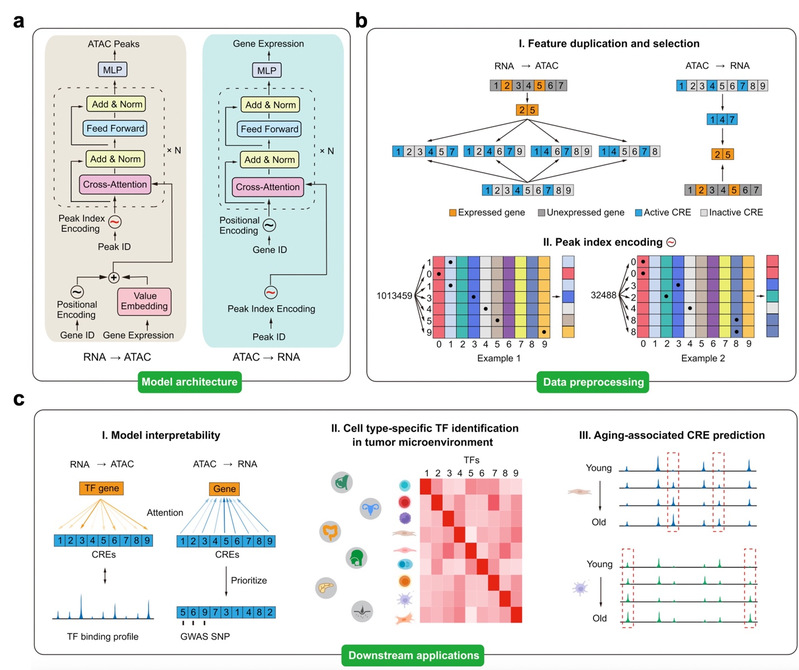
图1 Cisformer的模型框架
研究人员通过多项测试发现,Cisformer在不同来源的单细胞多组学数据中表现出优异的数据生成能力与模型可解释性。当利用单细胞转录组数据生成表观组数据时,相较于BABEL和scButterfly,Cisformer生成的单细胞染色质可及性数据更好地保留了细胞类型特异性。此外,Cisformer通过模型内在的注意力机制,揭示了不同类型的基因对染色质可及性调控强度的差异。当利用单细胞染色质可及性数据生成转录组数据时,Cisformer预测得到的基因表达谱比ArchR和SCARlink更接近真实数据,同时能更好地建立非编码元件与其靶基因的关联(图2)。
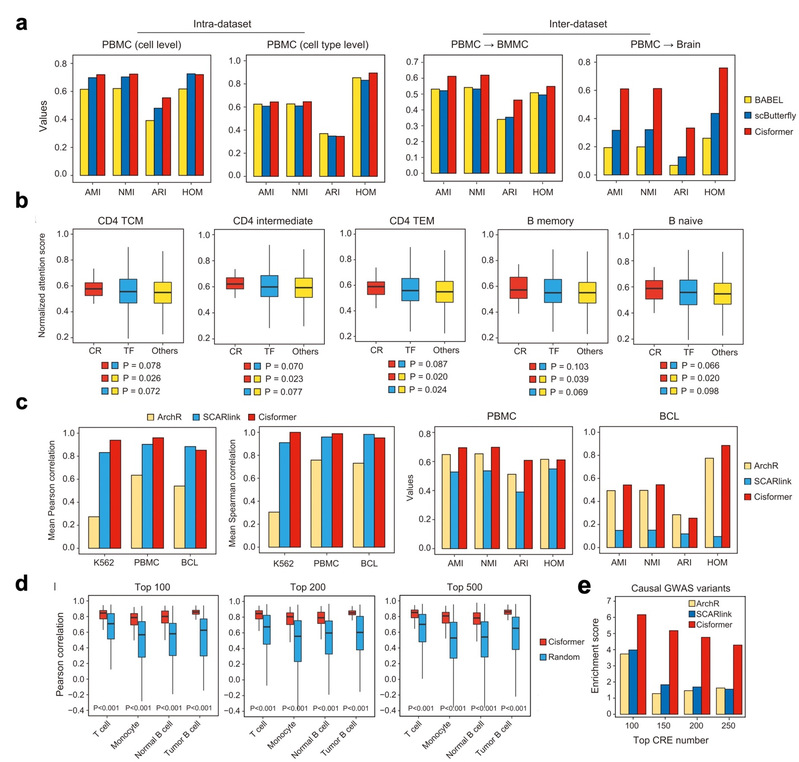
图2 Cisformer具备优异的跨模态数据生成能力与模型可解释性
此外,研究人员进一步将Cisformer应用于肿瘤发生与器官衰老这两个重要的生物学过程中。Cisformer在利用单细胞染色质可及性数据生成基因表达信息后,通过注意力机制分别从人类CD8+ T细胞、成纤维细胞以及巨噬细胞中识别出亚型特异的转录因子,其中部分与相关文献报道相符。而针对小鼠肾脏衰老过程,Cisformer通过单细胞转录组数据生成染色质可及性数据,鉴定出了衰老相关肾脏细胞特异与共同的潜在调控元件与转录因子(图3)。
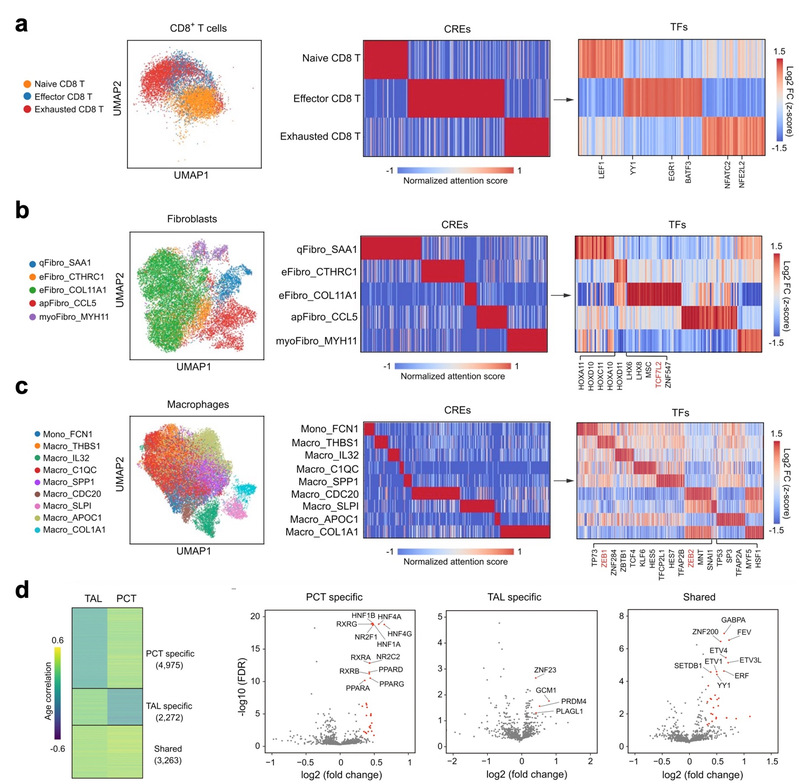
图3 Cisformer识别癌症发生与器官衰老相关的潜在调控元件与转录因子
综上,该项研究不仅证明了基于注意力机制的语言模型在单细胞跨模态生成任务中的适用性,还揭示了生理与病理状态下转录组与表观组相互作用的分子机制,为癌症的临床诊疗与器官衰老的有效干预提供了重要的研究视角与理论指导。
同济大学生命科学与技术学院/附属同济医院/上海梧桐岛生命科学研究院王晨飞教授为该论文的通讯作者,博士后季鲁章与博士生邹启航为共同第一作者。该研究得到了科技部、国家自然科学基金委、中国博士后科学基金会与上海市科委等项目的重要支持。
参考文献
1. Ma S, Zhang B, LaFave LM, Earl AS, Chiang Z, Hu Y, et al. Chromatin potential identified by shared single-cell profiling of RNA and chromatin. Cell. 2020;183(4):1103–16.
2. Wu KE, Yost KE, Chang HY, Zou J. Babel enables cross-modality translation between multiomic profiles at single-cell resolution. Proc Natl Acad Sci U S A. 2021;118(15):e2023070118.
3. Zhang R, Meng-Papaxanthos L, Vert JP, Noble WS. Multimodal single-cell translation and alignment with semi- supervised learning. J Comput Biol. 2022;29(11):1198–212.
4. Cao Y, Zhao X, Tang S, Jiang Q, Li S, Li S, et al. Scbutterfly: a versatile single-cell cross-modality translation method via dual-aligned variational autoencoders. Nat Commun. 2024;15(1):2973.
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


