EMBO molecular medicine|边杉/郭贞明/湖南省儿童医院王华/湖南省妇幼毛翛合作发现小头畸形又一致病基因——CETN3
原发性小头畸形(primary microcephaly)是一种相对罕见的疾病,其特征是患者头围的显著减小,主要致病原因是神经干细胞基因表达异常,从而影响了正常的神经发生过程,导致神经元数量的减少[1, 2]。目前已知有32个基因与原发性遗传性小头畸形 (microcephaly primary hereditary, MCPH)相关,包括MCPH1、ASPM、WDR62等[3-5],大多数MCPH基因参与调节细胞周期、中心体组装、DNA修复和细胞凋亡等生物学过程[6]。
近日,同济大学边杉/郭贞明/湖南省儿童医院王华/湖南省妇幼毛翛课题组在临床上发现了一个携带CETN3缺失型突变的小头畸形病人,该患者的头围比正常水平小2-3个标准差。但是到目前为止,尚未清楚CETN3在人类大脑皮层发育中的调控功能。于是,研究人员们合作对该基因对大脑皮层发育的调控功能进行了研究,并在EMBO molecular medicine上发表了题为CETN3 Deficiency Induces Microcephaly by Disrupting Neural Stem/Progenitor Cell Fate through Impaired Centrosome Assembly and RNA Splicing的研究文章。
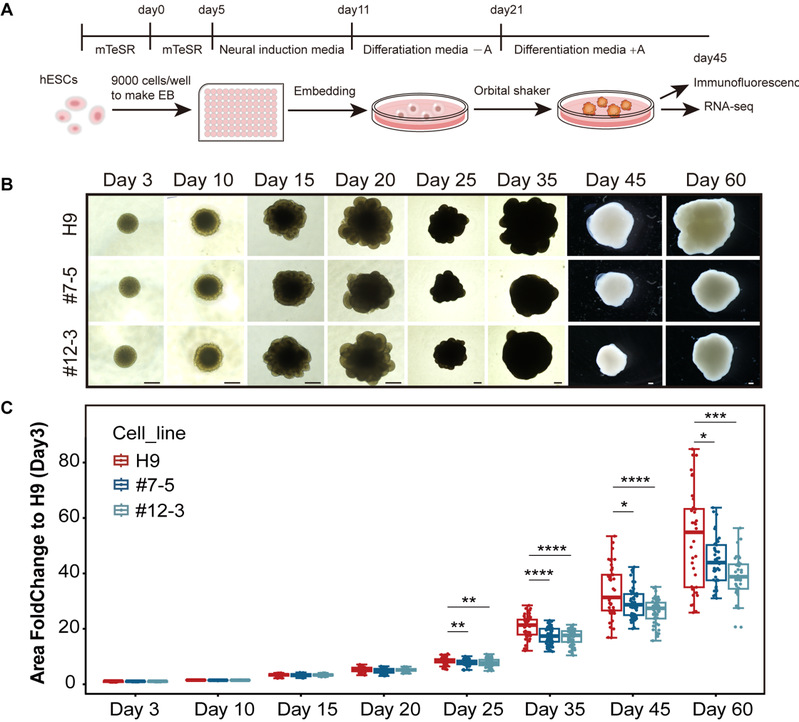
图1 CETN3缺失的人脑类器官表现出体积的减小
Centrin蛋白属于钙结合蛋白EF-hand超家族,含有四个钙结合结构域,在细胞中主要存在于与微管组织中心相关的结构中,例如中心体、纤毛或鞭毛、纺锤体等,大部分centrin蛋白在细胞质和细胞核中也有分布[7]。Centrin蛋白在进化上高度保守,但不同的生物含有的该蛋白编码基因数量不同,小鼠中有四个(Cetn1-4),而人类含有三个(CETN1-3)[8, 9]。这些蛋白的表达模式在不同的组织和细胞中有所差异,暗示不同的centrin蛋白可能参与了不同的生物学过程[10-12]。
文章中,作者以探究CETN3在大脑皮层发育中的作用为重点,鉴定CETN3缺失突变导致小头畸形的致病可能性,并阐明其致病分子机制。首先,他们检测了CETN3在大脑皮层中的表达,发现其广泛表达于各种类型的细胞中。接着,他们利用CRISPR-Cas9和Cre-LoxP基因编辑技术构建了CETN3敲除的人胚胎干细胞(hESC)系和CETN3突变的诱导多能干细胞(iPSC)系,并以这些细胞系为基础,通过2D培养获得了神经干/祖细胞(NS/PC),通过3D培养获得大脑类器官。该类器官模型重现了病人中大脑体积减小的表型,证实了CETN3的致病性(图1)
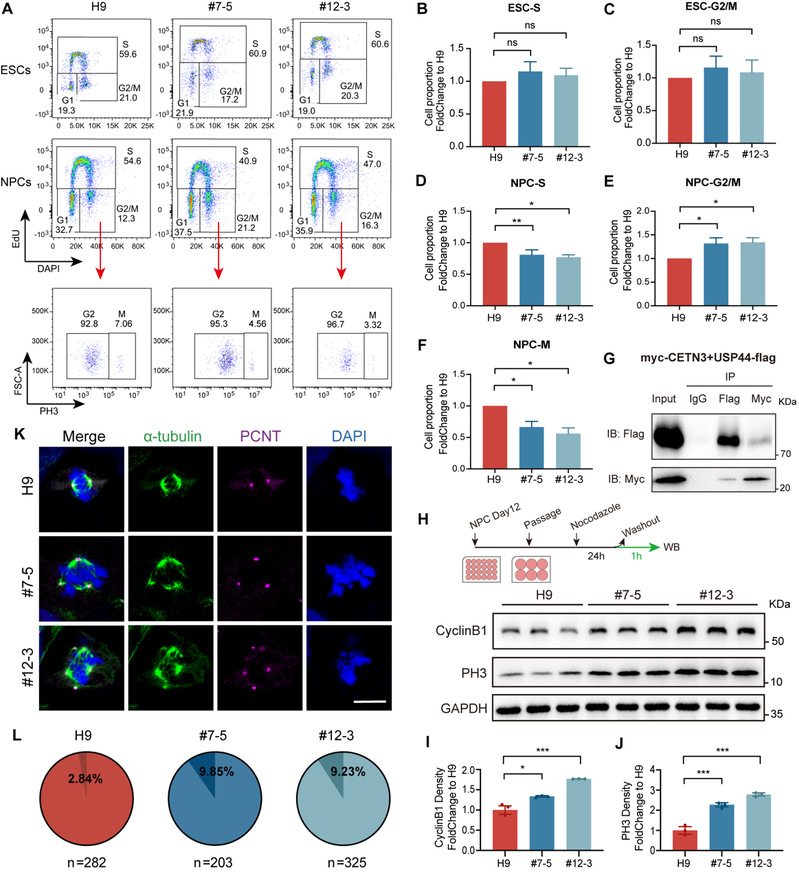
图2 CETN3缺失影响了NS/PC的细胞周期和中心体复制
接着,利用RNA-seq、免疫荧光染色、流式细胞术等方法,作者团队进一步对CETN3蛋白调控神经发育的分子机制进行了研究,并发现CETN3通过调节NS/PC增殖、分化、凋亡和RNA剪接来影响神经发生,具体如下:(1)CETN3缺失引起了NS/PC有丝分裂纺锤体方向的改变,使其更倾向分化而不是增殖;(2)CETN3缺失导致NS/PC细胞周期受损,从而影响细胞增殖;(3)CETN3缺失引发中心体过度复制,导致NS/PC凋亡(图2);(4)CETN3还可以通过参与RNA剪接来间接影响与中心粒、纤毛和细胞周期相关的基因的表达(图3)。值得关注的是,这是首次发现CETN3在调节RNA剪接方面的功能,作者团队还鉴定出了CETN3的这一功能可能与USP49的去泛素化功能和核小体H2B的泛素化水平有关。
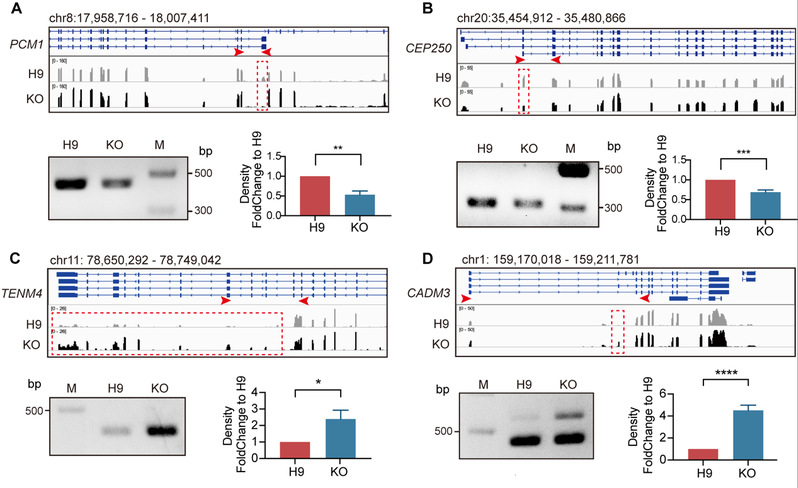
图3 CETN3缺失影响了NS/PC的RNA剪接
最后,作者还构建了Cetn3前脑特异性敲除的小鼠,以探究其在其他哺乳类神经发育中是否有相似的功能。结果发现小鼠中表型比较微弱,暗示CETN3在不同物种中的功能可能存在差异。
综上所述,该文章以大脑类器官为主要模型,首次揭示了CETN3在哺乳动物神经发育中的作用及其背后的分子机制。本研究证明了CETN3通过调节纺锤体定向、细胞周期、中心体复制等生物学过程参与了NPCs增殖和分化之间的平衡调控;本研究还首次发现CETN3通过与USP49相互作用参与了NPCs中的RNA剪接过程,从而间接影响了细胞周期和神经发育(图4)。这些结果提示了CETN3在人类大脑新皮层发育中的重要性,扩展了我们对原发性小头畸形致病基因的认识,为后续的临床检测和疾病治疗提供了新见解。

图4 CETN3缺失通过多种途径影响神经发育
参考文献:
[1] Becerra-Solano L E, Mateos-Sánchez L, and López-Muñoz E, Microcephaly, an etiopathogenic vision[J]. Pediatr Neonatol,2021,62(4):354-360.
[2] Pirozzi F, Nelson B, and Mirzaa G, From microcephaly to megalencephaly: determinants of brain size[J]. Dialogues Clin Neurosci,2018,20(4):267-282.
[3] Lim Y, Transcription factors in microcephaly[J]. Front Neurosci,2023,17:1302033.
[4] Asif M, Abdullah U, Nürnberg P, et al., Congenital Microcephaly: A Debate on Diagnostic Challenges and Etiological Paradigm of the Shift from Isolated/Non-Syndromic to Syndromic Microcephaly[J]. Cells,2023,12(4).
[5] Farcy S, Hachour H, Bahi-Buisson N, et al., Genetic Primary Microcephalies: When Centrosome Dysfunction Dictates Brain and Body Size[J]. Cells,2023,12(13).
[6] Naveed M, Kazmi S K, Amin M, et al., Comprehensive review on the molecular genetics of autosomal recessive primary microcephaly (MCPH)[J]. Genet Res (Camb),2018,100:e7.
[7] Paoletti A, Moudjou M, Paintrand M, et al., Most of centrin in animal cells is not centrosome-associated and centrosomal centrin is confined to the distal lumen of centrioles[J]. J Cell Sci,1996,109 ( Pt 13):3089-3102.
[8] Moretti E, Noto D, Corsaro R, et al., Focus on centrin in normal and altered human spermatozoa[J]. Syst Biol Reprod Med,2023,69(3):175-187.
[9] Martinez-Sanz J and Assairi L, New insights into the interaction of centrin with Sfi1[J]. Biochim Biophys Acta,2016,1864(4):319-330.
[10] Ying G, Frederick J M, and Baehr W, Deletion of both centrin 2 (CETN2) and CETN3 destabilizes the distal connecting cilium of mouse photoreceptors[J]. J Biol Chem,2019,294(11):3957-3973.
[11] Dantas T J, Wang Y, Lalor P, et al., Defective nucleotide excision repair with normal centrosome structures and functions in the absence of all vertebrate centrins[J]. J Cell Biol,2011,193(2):307-318.
[12] Gavet O, Alvarez C, Gaspar P, et al., Centrin4p, a novel mammalian centrin specifically expressed in ciliated cells[J]. Mol Biol Cell,2003,14(5):1818-1834.
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


