Nature Methods |刘琦教授团队开发单细胞多模态整合算法的全面基准评估平台
近年来,单细胞多模态组学技术取得突破性进展,科学家已能在单细胞分辨率下实现对基因组、转录组、蛋白质组及空间组学的多维度联合解析。这一技术突破使得研究者能够通过单细胞尺度的多组学定量,系统构建细胞在发育分化、疾病演进等过程中的全维度分子图谱。近年来公开的海量单细胞多模态数据集,使得基于多组学数据构建细胞状态预测大模型成为可能。面对日益增长的多模态数据,如何有效整合不同分子模态以及如何处理海量异构数据来解释细胞的复杂调控机制,已成为当前单细胞多模态研究领域的核心挑战。
同济大学生命科学与技术学院-上海自主智能无人系统科学中心刘琦教授交叉团队长期致力于组学智能解析及精准医学应用,前期在单细胞组学驱动的虚拟细胞构建AI方法路径,特别是跨组学整合的AI算法层面进行了系统而创新性的探索,曾开发了基于隐空间联合嵌入的单细胞RNA-seq和ATAC-seq整合算法scMVP[1],以及面向低质资源数据的T细胞转录组和TCR跨模态整合算法UniTCR[2]。近期,刘琦教授团队在Nature Methods杂志发表了题为“Benchmarking single-cell multi-modal data integrations ”的论文,发布了面向单细胞多模态整合的全面基准评估平台SCMMIB(Single Cell Multi-Modal Integration Benchmark),旨在为领域内的单细胞多模态整合算法提供一个具备全面性、定量化、多尺度、多指标的系统评估计算平台。该平台的测试评估涵盖了40个软件所涉及的65种整合算法,包含了RNA和ATAC (DNA,高维度)、 ADT(蛋白,低维度)和空间组学。根据多模态数据类型和数据集配对关系,该平台为多模态整合算法设计了六大类基准评估任务(图1)。
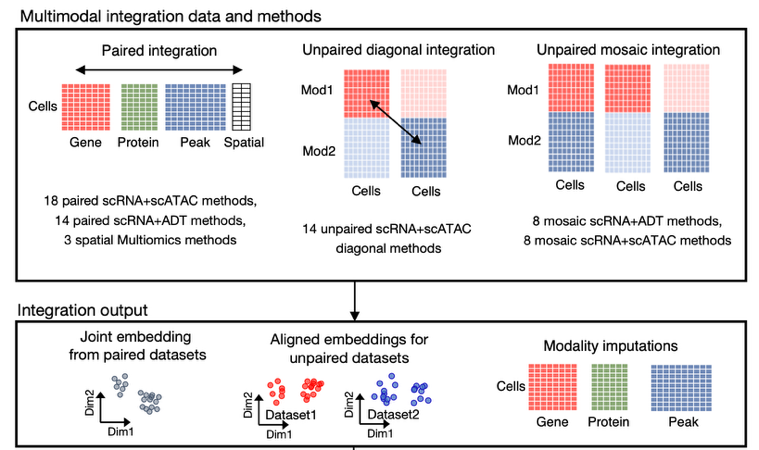
图1. SCMMIB平台框架设计
针对多模态整合不同应用场景、该平台构建了兼具可用性、准确性和稳定性的三维度基准评价指标和流程(图2)。其中,可用性指标主要针对不同数据集大小下(500到50万细胞)算法、硬件平台(仅CPU/GPU加速)下软件是否可以正常使用。准确性指标不依赖于多模态整合算法的数据金标准,评估了生物学结构保留、批次去除、和细胞对齐三类隐空间指标和跨模态生成准确性。稳定性指标主要衡量算法多次运行及不同模态数据集质量下算法性能和结果的稳定性。
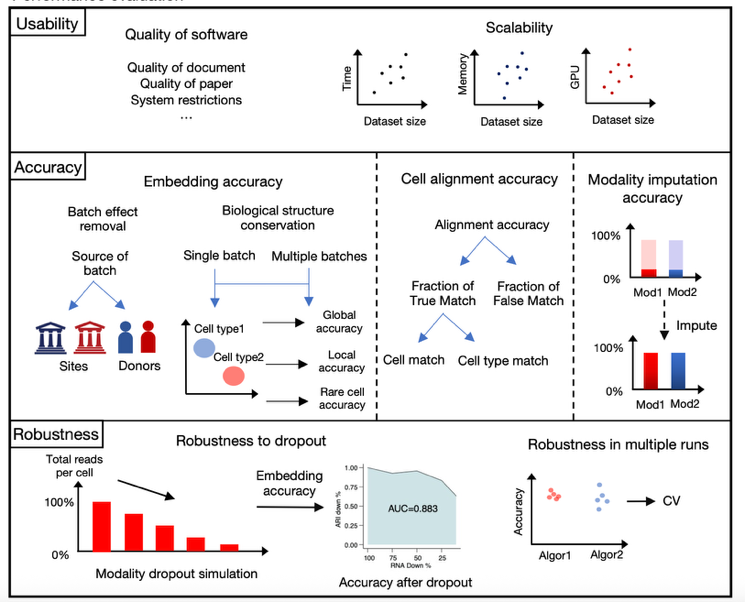
图2. SCMMIB平台评估流程
在模态匹配信息完全的配对(paired)多模态整合任务中,国际主流的Seurat 工具包中权重近邻(WNN)算法[3]在RNA+ATAC和RNA+ADT均获得了最好的综合性能,优于现有设计更为复杂的深度学习模型。
在部分或者全部匹配信息缺失的整合任务中,若干深度生成模型表现尤为突出;在部分模态信息匹配的马赛克整合(unpaired mosaic)任务中, MIDAS算法等[4]在隐空间准确性和稳定性上表现最佳,在跨模态生成(imputation)任务中MIDAS性能显著优于同类算法,并且作为无监督算法,其准确性已经接近多模态有监督(10%预测模态输入)生成模型(图3);在完全非配对(unpaired diagonal)整合任务中,GLUE算法[5]在模态对齐准确性的所有指标中领先其他算法,对齐性能已接近有部分配对信息马赛克整合算法;在空间多组学任务中,领域内已有算法相对于空转经典分析工具(Harmony[6], STAligner[7])并未有明显优势,仍有待发展创新算法对空间多组学数据整合和应用进行进一步探索。
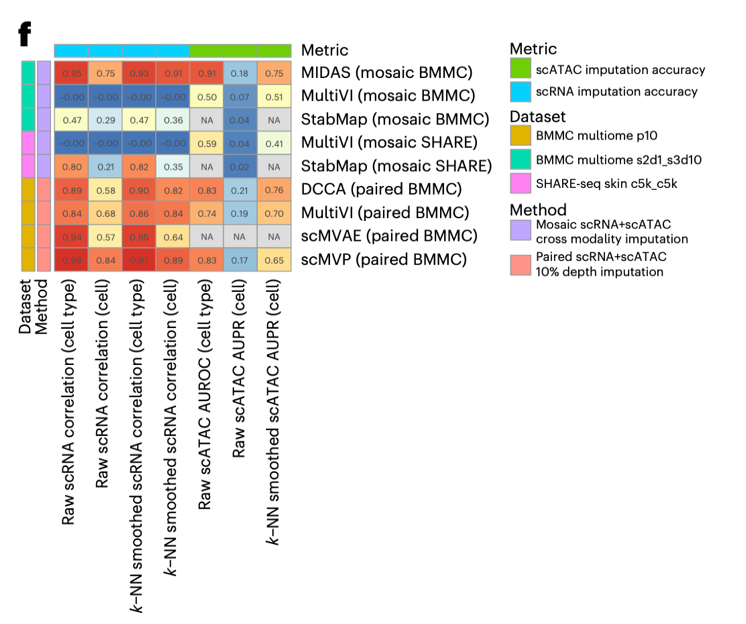
图3 马赛克整合算法和配对整合算法生成性能
同时,该平台还配套开发了一个用户友好的Web Server,提供完整的评估结果,并建立了一套标准化的计算流程,可帮助研究人员根据自身数据处理需求通过该计算流程选择最合适的整合方法,并用于新算法的性能自动验证。
综上所述,该平台对单细胞多模态整合领域的研究提供了一个系统而全面的基准评估框架,其评估结果为单细胞DNA、RNA、蛋白及空间组学数据的整合方法评估以及跨模态生物学知识智能发现提供了一个全面的参考和指南,有望进一步推动单细胞跨模态整合领域的AI方法学开发、细胞调控关系解析以及相关的生物学应用。
该论文第一作者是刘琦教授课题组的傅沙镠博士和博士生王曙光,通讯作者是同济大学刘琦教授。该研究得到了高亚威教授团队的大力支持。该研究成果获得国家自然科学基金青年科学基金项目(A类)、上海市计算生物学重点专项以及教育部前沿科学中心专项项目的资助。
参考文献:
1.Li G, Fu S, Wang S, Zhu C, Duan B, Tang C, Chen X, Chuai G, Wang P, Liu Q: A deep generative model for multi-view profiling of single-cell RNA-seq and ATAC-seq data. Genome Biology 2022, 23:20.
2.Gao Y, Dong K, Gao Y, Jin X, Yang J, Yan G, Liu Q: Unified cross-modality integration and analysis of T cell receptors and T cell transcriptomes by low-resource-aware representation learning. Cell Genom 2024, 4:100553.
3.Hao Y, Hao S, Andersen-Nissen E, et al: Integrated analysis of multimodal single-cell data. Cell 2021, 184:3573-3587 e3529.
4.He Z, Hu S, Chen Y, et al: Mosaic integration and knowledge transfer of single-cell multimodal data with MIDAS. Nat Biotechnol 2024.
5.Cao Z-J, Gao G: Multi-omics single-cell data integration and regulatory inference with graph-linked embedding. Nature Biotechnology 2022.
6.Korsunsky I, Millard N, Fan J, Slowikowski K, Zhang F, Wei K, Baglaenko Y, Brenner M, Loh PR, Raychaudhuri S: Fast, sensitive and accurate integration of single-cell data with Harmony. Nat Methods 2019, 16:1289-1296.
7.Zhou X, Dong K, Zhang S: Integrating spatial transcriptomics data across different conditions, technologies and developmental stages. Nat Comput Sci 2023, 3:894-906.
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


