Nature Communications | 高绍荣/康岚团队阐明RNA结合蛋白Lsm1在合子雌雄原核的组蛋白不对称分布和修饰中的作用机制
多细胞生物体的发育是一个系统过程,细胞在时间和空间上受到精确的细胞命运决定控制从而获得不同的身份。表观遗传调控与细胞命运决定的关系受到广泛重视,而小鼠植入前胚胎发育过程中的细胞命运和表观修饰的剧烈变化使其成为进行机制研究的绝佳模型。这一过程的表观遗传变化不是简单地发生在整体水平,而是受到精确控制,尤其是在合子阶段。受精后,第二极体排出,母源基因组逐步形成雌原核,来自精子的父源基因组经历鱼精蛋白与组蛋白的替换,形成雄原核[1, 2]。研究发现,受精后H3.3优先进入雄原核中,取代鱼精蛋白,而H3.1和H3.2在两个原核中都存在,但在雌原核中的含量显著高于雄原核[3-5],H3.3的缺失会影响着床前胚胎的正常发育[6],HIRA/DAXX–ATRX复合体和CAF-1复合体具有调控组蛋白H3变体掺入的作用[7-9],但雌雄原核间不对称H3变体的掺入机制,及其对受精卵表观遗传修饰建立的影响还未完全阐明。
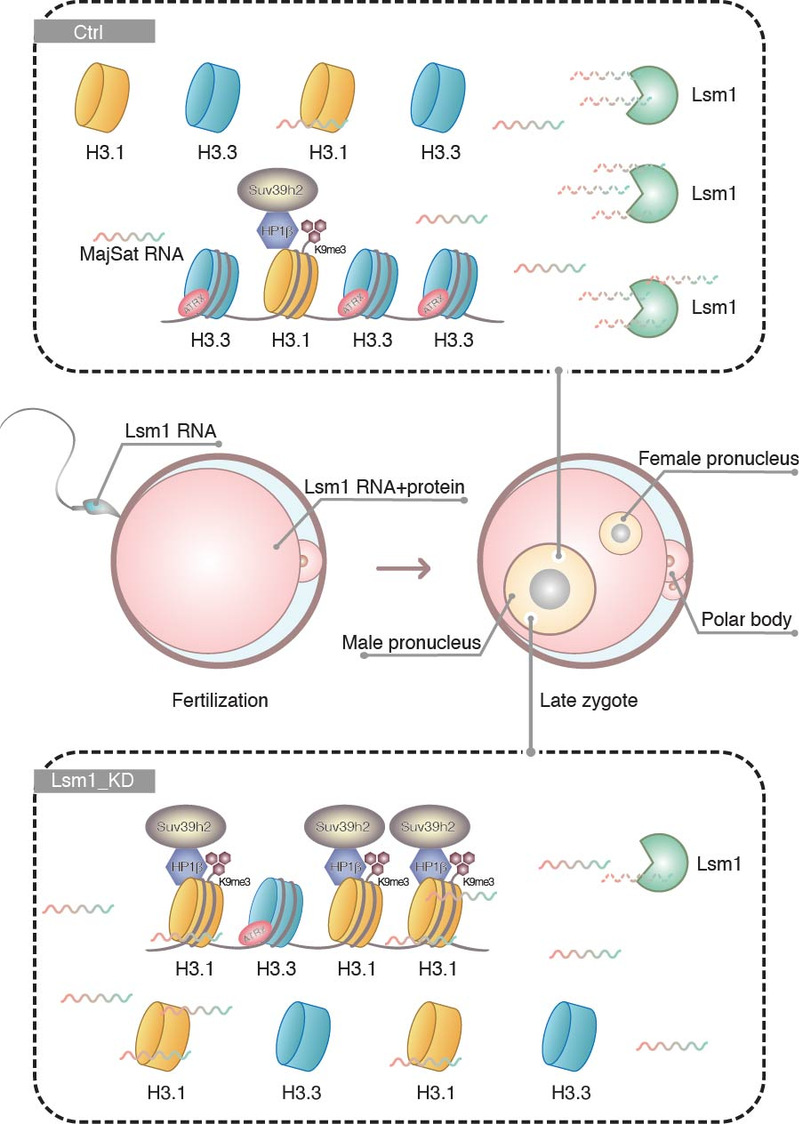
2023年2月21日,同济大学生命科学与技术学院高绍荣/康岚团队在Nature Communications在线发表了题为LSM1-mediated Major Satellite RNA decay is required for nonequilibrium histone H3.3 incorporation into parental pronuclei的研究论文, 该工作阐明了RNA结合蛋白Lsm1通过调节近着丝粒RNA(Major Satellite RNA)的降解,影响小鼠合子雌雄原核不对称组蛋白变体掺入以及表观修饰模式的建立。
在本项研究中,研究人员以课题组的早期胚胎发育蛋白质谱数据为基础,发现RNA降解相关蛋白Lsm1的敲降会导致合子非对称表观修饰模式的丧失--雄原核染色质出现H3K9me3修饰,并显著降低早期胚胎发育能力。在此基础上,利用total-RNA测序和免疫荧光染色发现,Lsm1通过调控组蛋白H3.1/H3.3蛋白定位而非表达来发挥作用。
研究人员利用微量细胞RIP-seq寻找合子中Lsm1直接作用分子,发现其结合并调控靶分子近着丝粒RNA(Major Satellite RNA)的降解。在此基础上,利用一系列功能实验证明近着丝粒RNA参与雌雄原核不对称H3K9me3修饰的调控,并影响早期胚胎发育,它的敲降可以逆转Lsm1敲降合子中异常的组蛋白掺入和修饰。近着丝粒RNA的过表达会造成雄原核中H3.1比例增加而H3.3比例减少,利用RNA pull-down进一步证明它对H3.1具有更强的亲和性。
最后,研究人员发现小鼠精子和卵子中均存在Lsm1的mRNA,细胞核中Lsm1的缺乏可能是造成体细胞核移植效率低下的原因之一。实验结果表明,过表达Lsm1可在一定程度上提高小鼠体细胞核移植胚胎发育能力。
综上所述,研究人员提出Lsm1介导的近着丝粒RNA降解调控了小鼠合子雌雄原核的组蛋白不对称分布和修饰的理论,进一步完善了早期胚胎表观修饰模式建立机制,对于理解细胞命运决定和全能性建立机制具有重要意义。
同济大学高绍荣教授和康岚教授为本文共同通讯作者,同济大学博士后朱江、博士生陈康,罗切斯特大学孙禹博士为共同第一作者,同济大学博士生叶文、刘军涛、张丹丹、苏楠等为本研究做出了重要贡献。该项工作得到了国家重点研发计划和国家自然科学基金等项目的重要支持。
文献:
1. Chazaud, C. and Y. Yamanaka, Lineage specification in the mouse preimplantation embryo. Development, 2016. 143(7): p. 1063-74.
2. Martin, C., et al., Genome restructuring in mouse embryos during reprogramming and early development. Dev Biol, 2006. 292(2): p. 317-32.
3. Akiyama, T., et al., Dynamic replacement of histone H3 variants reprograms epigenetic marks in early mouse embryos. PLoS Genet, 2011. 7(10): p. e1002279.
4. Santenard, A., et al., Heterochromatin formation in the mouse embryo requires critical residues of the histone variant H3.3. Nat Cell Biol, 2010. 12(9): p. 853-62.
5. Torres-Padilla, M.E., et al., Dynamic distribution of the replacement histone variant H3.3 in the mouse oocyte and preimplantation embryos. Int J Dev Biol, 2006. 50(5): p. 455-61.
6. Tang, M.C., et al., Contribution of the two genes encoding histone variant h3.3 to viability and fertility in mice. PLoS Genet, 2015. 11(2): p. e1004964.
7. Pchelintsev, N.A., et al., Placing the HIRA histone chaperone complex in the chromatin landscape. Cell Rep, 2013. 3(4): p. 1012-9.
8. Shibahara, K. and B. Stillman, Replication-dependent marking of DNA by PCNA facilitates CAF-1-coupled inheritance of chromatin. Cell, 1999. 96(4): p. 575-85.
9. Voon, H.P. and L.H. Wong, New players in heterochromatin silencing: histone variant H3.3 and the ATRX/DAXX chaperone. Nucleic Acids Res, 2016. 44(4): p. 1496-501.
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


