Cell Discovery |高绍荣/陈嘉瑜/张勇/李晓翠合作阐明组蛋白修饰调控人类原始生殖细胞发育关键事件的表观遗传机制
哺乳动物原始生殖细胞(Primordial germ cell,PGC)是生殖细胞的前体,最终发育产生高度特化的配子——精子和卵子。原始生殖细胞的正确发育是维持物种内遗传稳定性和连续性的必要条件,由此保证了遗传和表观遗传信息能忠实地从一代传递至下一代。原始生殖细胞在发育中经历了非常独特的事件,包括细胞的迁移、生殖嵴定位、性别决定、减数分裂和配子成熟等过程。更为重要的是,在这一进程中原始生殖细胞的转录组和表观遗传组发生了彻底而广泛的重编程,从而为后续配子发生和细胞全能性的建立做好了充分准备。然而,受限于有限的细胞数量和分子生物学技术方法,长久以来我们对人类 PGC(hPGC)发育过程中表观遗传组的确切变化知之甚少。
近年来,随着微量组学技术和相关生物信息学的发展,以北京大学乔杰院士和汤富酬教授为代表的多个国内外研究团队对hPGC的转录组、DNA甲基化组和染色质可及性等的动态变化规律进行了深入探究1-7。研究发现,男性和女性hPGC 在发育中呈现逐步的基因表达变化规律,并在细胞迁移、有丝分裂、减数分裂和配子发生中表现出异质性。同时,hPGC中的染色质可及性和 DNA 甲基化状态与处于相应发育阶段的小鼠相似,表明两个物种在原始生殖细胞的重编程动态变化上的保守性。然而,目前对hPGC中组蛋白修饰的研究主要基于免疫荧光染色,其全基因组分布、动态变化规律以及与其他表观遗传修饰如DNA甲基化和染色质可及性的相关性尚不清楚。此外,它们是否参与hPGC全基因组低甲基化下的转录调控和特定发育事件如X染色体再激活和DNA去甲基化逃逸等仍有待回答。
2023年2月3日,同济大学高绍荣教授、陈嘉瑜教授、张勇教授与同济大学附属第一妇婴保健院李晓翠教授团队合作,在Cell Discovery杂志(IF: 38)在线发表了题为“Resetting Histone Modifications During Human Prenatal Germline Development”的研究论文。该工作揭示了在长达数月的人原始生殖细胞染色质重编程中,三种核心组蛋白修饰(H3K4me3、H3K27me3和H3K9me3)在全基因组低甲基化状态下,协同调控基因和逆转座子表达、X染色体再激活和DNA去甲基化逃逸等事件的表观遗传机制。
在本项目研究中,研究人员利用微量细胞ULI-NChIP-seq技术,首次绘制了妊娠7周至23周发育期间内男性和女性原始生殖细胞中三个关键组蛋白修饰(H3K4me3、H3K27me3和H3K9me3)的高分辨率全基因组动态图谱(图1A)。研究表明 H3K4me3 表现出典型的启动子富集模式,与DNA甲基化所不同的是,H3K4me3修饰显示出与基因转录及染色质开放状态的高度相关性(图1B和1C)。相比而言,hPGC发育中H3K27me3 修饰呈现出相对低的全基因组富集度,且其主要定位于H3K4me3标记的区域并与染色质开放呈现负相关,在调控发育相关的特定二价启动子中发挥着重要作用(图1B和1C)。有意思的是,过去的研究表明女性胚胎及生殖细胞发育中存在独特的X染色体调控机制,称为X染色体抑制(X-chromosome dampening,XCD)8。其中,X染色体表现出不完全的再激活特征,受到lincRNA XACT的调控,而非依赖于传统lincRNA XIST的作用。本研究发现,XACT和XIST的表达在7-23 周男女hPGC中没有显著差异,且男女X染色体上的DNA甲基化也没有显著差异,提示它们均不能充分解释女性hPGC中X染色体再激活的调控。本研究发现相比于男性X染色体,女性X染色体上高度富集H3K4me3修饰,提示X染色体的再激活需要这些活跃的组蛋白修饰标记。考虑到女性hPGC的整体低甲基化状态和H3K4me3信号在X染色体上的富集,为何两条X染色体的激活只导致了基因表达增加了1.6倍,而不是2倍呢?2进一步的研究发现,与同期发育阶段的男性hPGC相比,女性hPGC在X染色体上表现出更高的H3K27me3和H3K9me3修饰水平,提示抑制性组蛋白修饰在限制女性hPGC 的X 染色体完全再激活中发挥着重要作用。
值得注意是,hPGC虽然在发育的早期(妊娠4周开始)就表现出DNA去甲基化,但是有一些特殊的区域始终呈现较高的甲基化水平,它们被称为去甲基化逃逸区域。本研究发现H3K9me3主要标记于这些逃逸区域,而不是发生去甲基化的区域。此外,异染色质修饰H3K27me3可能在这些逃逸区域上,发挥着协同巩固甲基化的作用。在发育早期,这些去甲基化逃逸区域上就可以同时检测到组蛋白H3K9me3和H3K27me3的共标记,表明这两个核心组蛋白标记足以在发育中持续保护这些逃逸区域不发生彻底的去甲基化,而此时全基因组仍在发生持续而彻底的去甲基化进程。相比而言,去甲基化区域很少同时出现H3K9me3和H3K27me3的共标记,且其表达量也相对较高。鉴于原始生殖细胞在发育中全局的 DNA 去甲基化和 H3K4me3 对基因表达的协同激活效应,本研究揭示了抑制性组蛋白修饰H3K9me3和H3K27me3共同参与hPGC中去甲基化逃逸区域的稳定维持与转录调控。值得一提的是,近日英国剑桥大学Surani课题组在Science Advances发表了题为“Epigenetic resetting in the human germ line entails histone modification remodeling”的研究工作也发现了抑制性组蛋白修饰H3K9me3和H3K27me3在全基因组低甲基化状态下对hPGC逆转座子原件的重要调控作用9。
同济大学陈嘉瑜教授、张勇教授和高绍荣教授为本文共同通讯作者,高睿助理教授、博士生曾诗扬和杨东旭及李晓翠教授为共同第一作者,刘文强教授、高亚威教授、柏丹丹博士、张林凤博士、陈川博士、洪玮主治医师和王蓓颖副主任医师等为本研究做出了重要贡献。该项工作得到了国家自然科学基金委和国家重点研发计划等项目的重要支持。
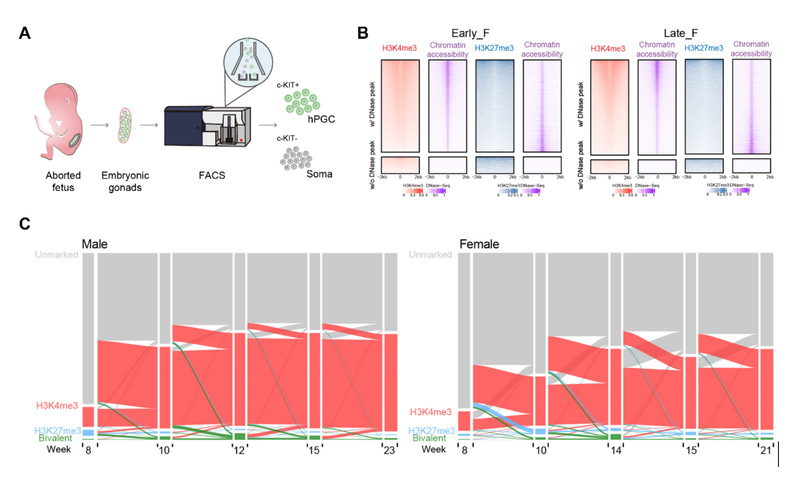
图1. 人原始生殖细胞组蛋白修饰的动态变化研究
(A) ULI-NChIP-seq分析的hPGC(c-KIT阳性)和性腺体细胞(soma) (c-KIT阴性)的流程图。(B) 早期和晚期女性hPGC中启动子上组蛋白修饰H3K4me3和H3K27me3及DNase-seq定义的染色质可及性的相关性热图。 (C) 冲积图显示H3K4me3(红色)-,H3K27me3(蓝色)-,二价修饰(绿色)-和未标记(灰色)-启动子在男性与女性hPGC发育中的动态变化规律。
文献
Gkountela, S. et al. DNA Demethylation Dynamics in the Human Prenatal Germline. Cell 161, 1425-1436, doi:10.1016/j.cell.2015.05.012 (2015).
Guo, F. et al. The Transcriptome and DNA Methylome Landscapes of Human Primordial Germ Cells. Cell 161, 1437-1452, doi:10.1016/j.cell.2015.05.015 (2015).
Guo, H. S. et al. DNA methylation and chromatin accessibility profiling of mouse and human fetal germ cells. Cell Res 27, 165-183, doi:10.1038/cr.2016.128 (2017).
Li, J. Y. et al. Accurate annotation of accessible chromatin in mouse and human primordial germ cells. Cell Res 28, 1077-1089, doi:10.1038/s41422-018-0096-5 (2018).
Li, L. et al. Single-Cell RNA-Seq Analysis Maps Development of Human Germline Cells and Gonadal Niche Interactions. Cell Stem Cell 20, 858-+, doi:10.1016/j.stem.2017.03.007 (2017).
Tang, W. W. C. et al. A Unique Gene Regulatory Network Resets the Human Germline Epigenome for Development. Cell 161, 1453-1467, doi:10.1016/j.cell.2015.04.053 (2015).
Li, L. et al. Dissecting the epigenomic dynamics of human fetal germ cell development at single-cell resolution. Cell Res 31, 463-477, doi:10.1038/s41422-020-00401-9 (2021).
Chitiashvili, T. et al. Female human primordial germ cells display X-chromosome dosage compensation despite the absence of X-inactivation. Nat Cell Biol 22, 1436-+, doi:10.1038/s41556-020-00607-4 (2020).
Gruhn, W. H. et al. Epigenetic resetting in the human germ line entails histone modification remodeling. Sci Adv 9, eade1257, doi:10.1126/sciadv.ade1257 (2023).
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


