National Science Review | 高亚威/吴悠/高绍荣研究团队揭示NEXT复合物核心成员ZCCHC8在精子发生过程中调控转座子LINE1的沉默机制

转座元件(Transposon Elements, TEs)是哺乳动物基因组的重要组成部分,其类型繁多,功能复杂,近年来的研究表明,TE可影响基因转录活性和染色质状态[1, 2]。在雄性小鼠生殖细胞中,TE的过度激活通常导致生殖缺陷,如减数分裂障碍和无精症。因此,TE活性的调控机制一直都是雄性生殖生物学的研究热点。在精子发生过程中TE活性的调控机制多样,包括从头DNA甲基化,抑制性组蛋白修饰(如H3K9me2/3),以及由PIWI蛋白及其相关piRNA介导的转录后或转录沉默等[3-5],但是否存在其他未知的转录后调控机制仍有待探索。
RNA外切体(RNA Exosome)是细胞中控制RNA降解和质量的重要复合物,被称为“细胞监视器”[6, 7]。在哺乳动物细胞中,NEXT复合物(Nuclear Exosome Targeting Complex,NEXT)被认为是RNA外切体的接头因子之一,主要负责靶向核内非polyA尾的RNA并引导RNA外切体进行降解。NEXT复合物包括RNA识别蛋白RBM7,锌指蛋白ZCCHC8以及RNA解旋酶hMTR4[8]。2019年高绍荣课题组的研究首次发现,NEXT复合物关键因子ZCCHC8在转座子LINE1 RNA降解中发挥重要作用,对于小鼠卵母细胞成熟、早期胚胎发育以及胚胎干细胞的功能都非常重要[9](Cell Reports | 高绍荣/高亚威团队首次发现NEXT复合物关键因子Zcchc8对LINE1 RNA的降解作用),而NEXT复合物在小鼠精子发生过程中的具体作用尚未被明确。
2024年12月17日,同济大学生命科学与技术学院的高亚威/吴悠/高绍荣研究团队在National Science Review上发表题为“Core Factor of NEXT Complex, ZCCHC8, Governs the Silencing of LINE1 during Spermatogenesis” 的研究论文,该研究揭示了ZCCHC8在精子发生过程中发挥的重要功能,提出了其对LINE1 RNA的靶向结合,进而影响染色质环境,协助减数分裂过程中染色质凝聚的调控机制。
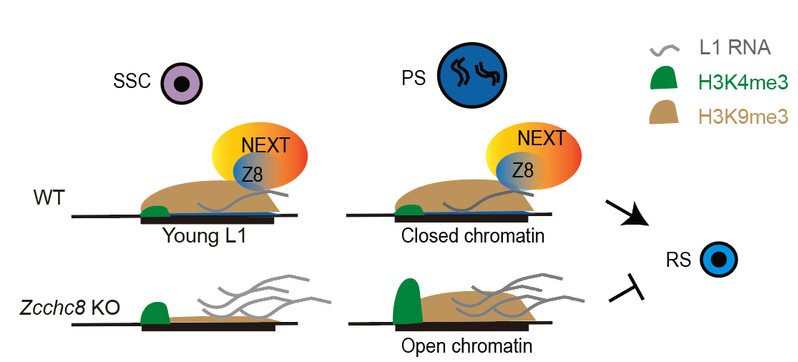
研究人员首先发现ZCCHC8基因敲除(Knockout, KO)的雄性小鼠表现出不育,精子数量减少且形态异常,圆形精子细胞数量下降,并伴随细胞凋亡增加。通过对第一波精子发生过程检测,发现出生后6天小鼠睾丸中的精原干细胞(THY1+细胞,Spermatogonial Stem Cell,SSC)数量明显减少,并出现第一波精子发生延迟的现象,说明ZCCHC8在精子发生初期,对精原干细胞和减数分裂进程具有重要作用。此前已有报道表明ZCCHC8可以结合TE RNA,在小鼠胚胎干细胞中偏向结合年轻LINE1 RNA,调控LINE1 RNA的降解。随后,研究人员对生精细胞中LINE1的RNA和蛋白水平进行进一步检测,发现在KO小鼠中精原干细胞和减数分裂期精母细胞中都有显著增加,其中年轻的LINE1亚型L1Md_A明显上调,且不依赖piRNA通路。紧接着,研究人员在出生6天小鼠的睾丸和成年小鼠的粗线期精母细胞(Pachytene Spermatocytes, PS)中进行了RNA免疫共沉淀(RIP-seq)实验,结果显示上调的LINE1亚型多被ZCCHC8靶向。进一步研究表明,ZCCHC8基因敲除小鼠的精原干细胞中H3K9me3修饰减少,而PS中H3K4me3修饰增加,这些变化也主要集中于年轻的LINE1亚型L1Md_A。由于异常升高的H3K4me3修饰通常与染色质开放程度偶联,减数分裂期精母细胞的染色质状态可能影响圆形精子(RS)形成过程中的染色质凝集,研究人员对PS和RS的染色质可及性(ATAC-seq)进行了分析,结果显示ZCCHC8缺失的PS和RS中LINE1区域的染色质可及性显著增加,表明ZCCHC8通过调控LINE1 RNA水平影响圆形精子形成过程中的染色质凝聚,揭示了NEXT复合物对于减数分裂和精子成熟的重要作用。
本研究首次证明了ZCCHC8在精子发生过程中直接结合年轻的L1 RNA,调控其RNA水平和基因组位点上的染色质状态,协助减数分裂过程染色质凝集,揭示了RNA监控系统对于减数分裂和精子成熟的重要作用,为生精细胞中转座子沉默的调控方式提供了新的理论,为相关男性不育症的疾病机制研究提供了新思路。
同济大学博士生颜如霜, 中国科学技术大学附属第一医院齐美杰博士和之江实验室张鹏飞博士为本文的并列第一作者。同济大学高亚威教授,吴悠副教授,高绍荣教授为本文的共同通讯作者。南京医科大学沈彬研究员为本项目提供了重要支持。
参考文献:
1.Liang L, Cao C, Ji L et al. Complementary Alu sequences mediate enhancer-promoter selectivity. Nature. 2023; 619(7971): 868-875. doi: 10.1038/s41586-023-06323-x
2.Haws SA, Simandi Z, Barnett RJ et al. 3D genome, on repeat: Higher-order folding principles of the heterochromatinized repetitive genome. Cell. 2022; 185(15): 2690-2707. doi: 10.1016/j.cell.2022.06.052
3.Barau J, Teissandier A, Zamudio N et al. The DNA methyltransferase DNMT3C protects male germ cells from transposon activity. Science. 2016; 354(6314): 909-912. doi: 10.1126/science.aah5143
4.Zoch A, Auchynnikava T, Berrens RV et al. SPOCD1 is an essential executor of piRNA-directed de novo DNA methylation. Nature. 2020; 584(7822): 635-639. doi: 10.1038/s41586-020-2557-5
5.Iwasaki YW, Siomi MC, Siomi H. PIWI-Interacting RNA: Its Biogenesis and Functions. Annu Rev Biochem. 2015; 84: 405-433. doi: 10.1146/annurev-biochem-060614-034258
6.Schmid M, Jensen TH. Controlling nuclear RNA levels. Nat Rev Genet. 2018; 19(8): 518-529. doi: 10.1038/s41576-018-0013-2
7.Meola N, Domanski M, Karadoulama E et al. Identification of a Nuclear Exosome Decay Pathway for Processed Transcripts. Mol Cell. 2016; 64(3): 520-533. doi: 10.1016/j.molcel.2016.09.025
8.Lubas M, Christensen MS, Kristiansen MS et al. Interaction profiling identifies the human nuclear exosome targeting complex. Mol Cell. 2011; 43(4): 624-637. doi: 10.1016/j.molcel.2011.06.028
9.Wu Y, Liu W, Chen J et al. Nuclear Exosome Targeting Complex Core Factor Zcchc8 Regulates the Degradation of LINE1 RNA in Early Embryos and Embryonic Stem Cells. Cell Rep. 2019; 29(8): 2461-2472 e2466. doi: 10.1016/j.celrep.2019.10.055
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


