Bioinformatics|王晨飞课题组开发基于单细胞和空间多组学数据构建基因调控网络方法SCRIPro
转录调控因子(TR),包括转录因子(TF)和染色质调控因子(CR),通过招募转录起始复合物和调节染色质可及性在基因调控中发挥关键作用。TR与其靶基因形成的复杂基因调控网络(GRNs)在不同细胞环境中高度动态化。随着单细胞技术的进步,基于单细胞转录组或表观基因组数据的GRNs推断方法纷纷涌现,但这些方法往往忽略了真实的染色质可及性状态,无法在单细胞水平上实现GRN预测,并且大量依赖基序(motif)参考来识别潜在靶标,导致细胞类型特异性的丧失。为了解决这些限制,王晨飞课题组于2022年开发了SCRIP[1],通过整合大量TR ChIP-seq和基序参考集合,从scATAC-seq数据重建单细胞TR活性和GRNs。尽管如此,SCRIP的有效性受到scATAC-seq数据普遍性和质量的影响。近年来,单细胞多组学数据的发展催生了新工具,这些工具利用成对或整合的多组学数据作为输入,通过线性/非线性回归方法构建GRNs,但仍然高度依赖基序信息,准确性和覆盖范围有待进一步提高。此外,空间多组学技术的发展使得在复杂组织中进行细胞结构的精确分析以及细胞间的空间相互作用成为可能,显著增强了我们对特定微环境中基因调控的理解。然而,目前大多数预测TR活性的工具并未考虑细胞的空间位置,忽视了同一微环境内不同细胞或点位表达相似性的影响,导致基于空间转录组数据的TR预测不准确。
2024年7月18日,同济大学王晨飞课题组在Bioinformatics杂志上在线发表文章Single-cell and spatial multiomic inference of gene regulatory networks using SCRIPro,开发了基于单细胞及空间多组学数据构建基因调控网络的工具SCRIPro(https://github.com/wanglabtongji/SCRIPro)。

SCRIPro首先通过考虑表达及空间相似性,将临近细胞拟合成SuperCell来解决单细胞或空间多组学信号稀疏性的问题。其次,SCRIPro除了从Cistrome DB数据库[2]中收集基于实验的高质量TR ChIP-seq峰的全基因组位置信号,还收集了1252个人类TR和994个小鼠TR的基序组成全面完整的TR全基因组参考。最后,SCRIPro对表观基因组数据进行拟合敲除,得到的TR-靶基因的重要得分,结合TR-靶基因的表达得分来构建GRN。研究人员通过将其应用于多套数据集包括人类T细胞的单细胞CRISPR激活数据集、人类B细胞淋巴瘤、小鼠毛囊发育单细胞多组学数据、小鼠胚胎Stereo-seq数据集在连续发育时间点和P22小鼠大脑空间-ATAC-RNA数据等,展示了SCRIPro的鲁棒性和多功能性,也显示了SCRIPro在不同生物学背景下的卓越性能和实用性。
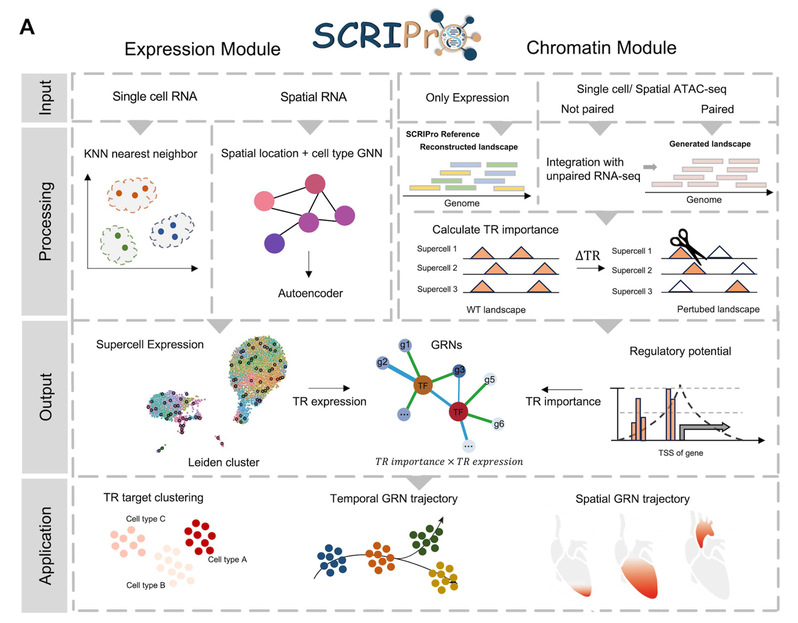
图1 SCRIPro 工作流程示意图
为了定量评估SCRIPro的性能,研究人员使用了一套已发表的单细胞CRISPR激活数据集,该数据集筛选了原代人T细胞中的T细胞刺激调节因子[3],作为GRN推断方法的基准(图2A)。我们比较了SuperCell方法的优越性以及确定了最佳分辨率(图2B-D),结果显示SCRIPro在AUROC和AUPRC指标上相较于现有的GRN推断方法SCING和SCENIC表现更加出色(图2E-F)。此外,通过比较不同的参考集,我们证明了SCRIPro采用更加完善的参考集不仅在预测准确性上具有明显优势,同时也在计算效率上表现更优(图2G)。总体而言,SCRIPro在多个评估指标上均展现了卓越的性能和效率。
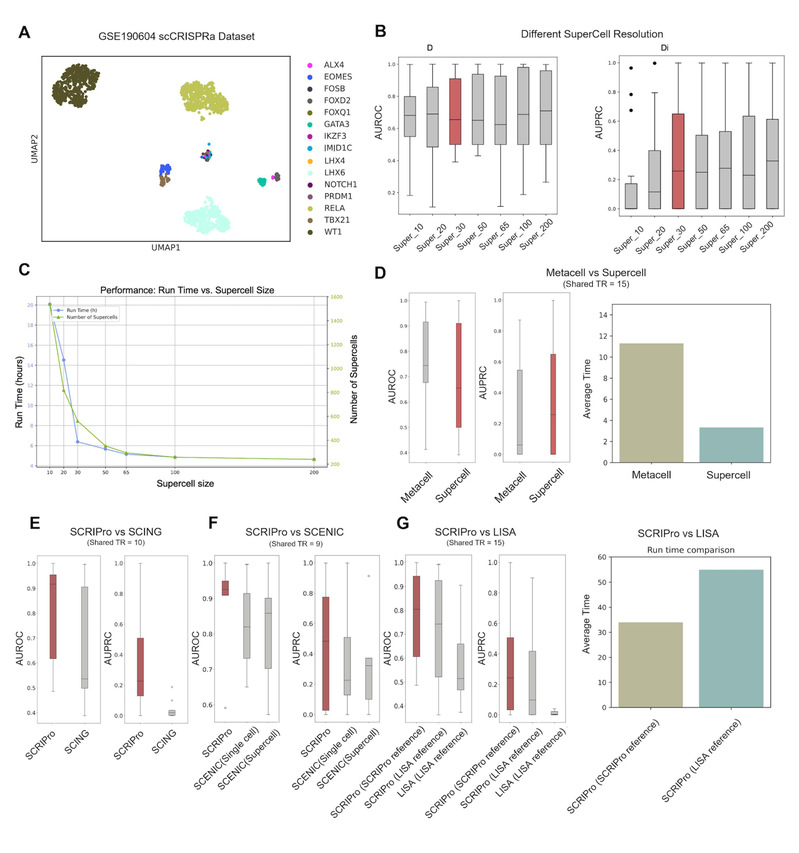
图2 SCRIPro 性能评估
为了证明SCRIPro也可以很好的应用于单细胞空间多组学数据集上,研究人员首先将E11.5到E15.5的小鼠胚胎发育的空间转录组数据进行配准(图3A-B)[4],并且使用SCRIPro识别了小鼠胚胎心脏发育过程中不同阶段特异的TRs(图3C),如Gata6、Jund、Mef2c等。研究人员进一步构建了基于心肌细胞的跨阶段GRN,并且按照E11.5到E13.5发育阶段定义了早期和晚期重要的调控因子如Nkx2-5、Clock等(图3D)。除了TR活性外,SCRIPro还稳健地识别了TR的下游靶基因,可用于评估不同TR的稳定或动态调控(图3E)。其中Prdm16的GRN在不同阶段表现出显著的动态变化,如E11.5阶段丢失的基因在糖原代谢中高度富集,而E13.5阶段的独特基因则特异性地富集于NADP代谢,提示Prdm16可能在心肌细胞的代谢重编程中发挥关键作用[5],这一过程与心肌细胞的增殖能力相关。总体而言,SCRIPro能够有效追踪和分析小鼠胚胎心脏发育过程中的TR及其调控网络,为空间转录组数据中新型谱系调控因子的发现奠定了基础。
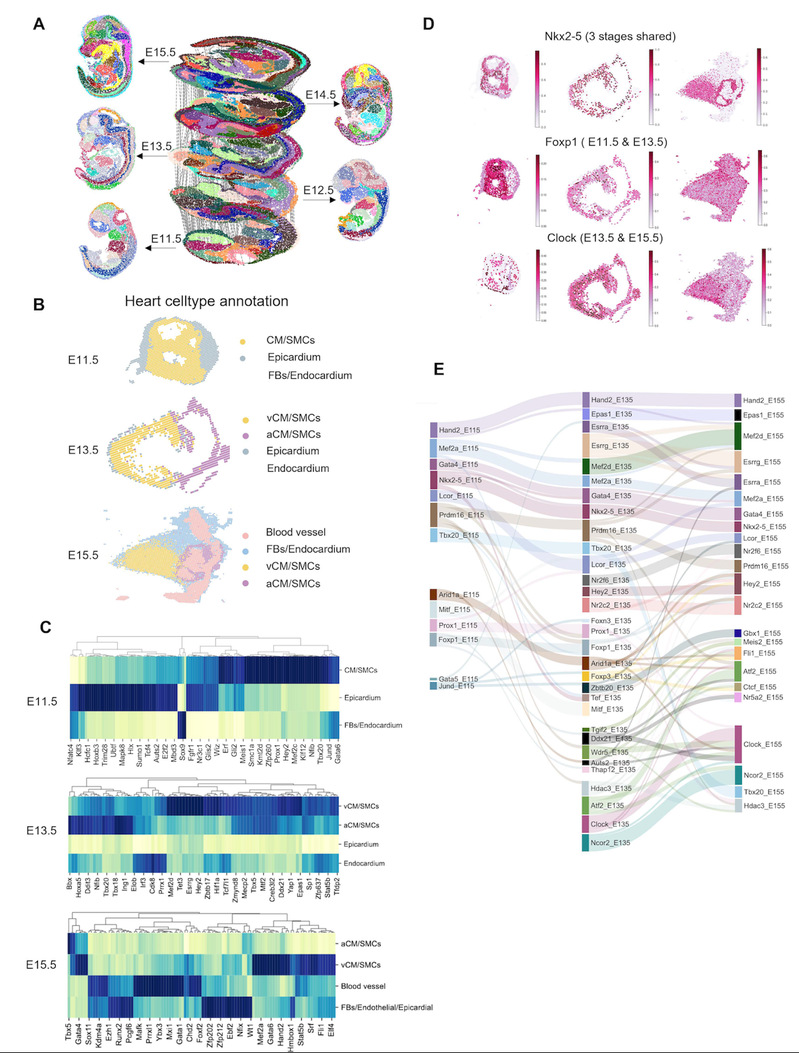
图3 SCRIPro应用于小鼠胚胎发育Stereo-seq空间转录组数据集
同济大学生命科学与技术学院王晨飞研究员为该论文通讯作者,同济大学生命科学与技术学院博士研究生常展赫、硕士研究生许云帆和董鑫博士为文章的共同第一作者,该项工作得到了生命学院高亚威教授、国家自然科学基金委及上海市科委等项目的重要支持。
1. Dong X, Tang K, Xu Y, Wei H, Han T, Wang C: Single-cell gene regulation network inference by large-scale data integration. Nucleic Acids Res 2022, 50:e126.
2. Zheng R, Wan C, Mei S, Qin Q, Wu Q, Sun H, Chen CH, Brown M, Zhang X, Meyer CA, Liu XS: Cistrome Data Browser: expanded datasets and new tools for gene regulatory analysis. Nucleic Acids Res 2019, 47:D729-d735.
3. Schmidt R, Steinhart Z, Layeghi M, Freimer JW, Bueno R, Nguyen VQ, Blaeschke F, Ye CJ, Marson A: CRISPR activation and interference screens decode stimulation responses in primary human T cells. Science 2022, 375:eabj4008.
4. Chen A, Liao S, Cheng M, Ma K, Wu L, Lai Y, Qiu X, Yang J, Xu J, Hao S, et al: Spatiotemporal transcriptomic atlas of mouse organogenesis using DNA nanoball-patterned arrays. Cell 2022, 185:1777-1792.e1721.
5. Cibi DM, Bi-Lin KW, Shekeran SG, Sandireddy R, Tee N, Singh A, Wu Y, Srinivasan DK, Kovalik JP, Ghosh S, et al: Prdm16 Deficiency Leads to Age-Dependent Cardiac Hypertrophy, Adverse Remodeling, Mitochondrial Dysfunction, and Heart Failure. Cell Rep 2020, 33:108288.
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


