同济大学生物信息系刘琦教授课题组在肿瘤新生抗原筛选计算领域取得系列进展
近日,同济大学生物信息系刘琦教授课题组成功开发了基于高通量测序数据进行肿瘤新生抗原排序筛选的计算方法pTuneos。相关研究论文《pTuneos: prioritizing tumor neoantigens from next-generation sequencing data》于10月31日在国际著名期刊Genome Medicine杂志在线发表【1】。

新生抗原是肿瘤细胞中的非同义突变经过转录翻译及加工得到的多肽。由于正常细胞中不会表达肿瘤新抗原,新抗原特异性免疫反应不会经历中枢耐受机制和外周耐受机制,所以理论上新抗原是具有广泛的治疗前景和临床应用价值的免疫治疗靶点。随着高通量测序技术的发展,全基因组和外显子测序技术可以帮助研究者获取基因组上的突变信息(包括点突变,插入缺失等),如何从这些数据中快速准确的鉴定候选新抗原并筛选出高免疫原性的新抗原肽是肿瘤免疫治疗领域一个亟待解决的问题。目前已有的工具方法如pVAC-seq,MuPeXI,neopepsee等一定程度上解决了新抗原鉴定的问题,但对候选新抗原的有效筛选和排序仍存在不足,主要包括:1)基于阈值筛选的方法过于粗糙,假阳性高;2)基于打分函数的方法所考虑的新抗原特征有限,无法准确评估其免疫原性;3)基于机器学习模型的方法特征未进行有效处理,所用训练数据不平衡性严重;4)模型运行速度较慢;5)已知模型往往基于体外试验模型数据训练,无法准确评估新抗原的体内真实免疫原性。
针对上述新抗原鉴定筛选过程中存在的问题,刘琦教授课题组提出了一个新颖有效的肿瘤新生抗原排序筛选计算框架pTuneos(prioritizing tumor neoantigens from next-generation sequencing data)(图一),用于从高通量测序数据中有效的预测新抗原并对其真实免疫原性进行评估和排序。该计算框架包含四个步骤:1)数据预处理。首先对外显子组测序数据和转录组测序数据进行质控并比对基因组,随后计算得到的基因突变谱,基因表达谱,HLA分型;2)候选新抗原鉴定。基于基因突变谱,基因表达谱,HLA分型计算得到未经筛选的候选新抗原列表;3)基于体外实验新抗原免疫活性数据训练特定机器学习模型并处理数据不平衡性,该模型可以初步筛选可被MHC复合物递呈和T细胞表面受体识别的新抗原;4)新抗原排序优化。即进一步基于新抗原产生、递呈和识别过程中的全面特征构建一种新抗原免疫原性排序优化函数,该打分函数可以对于第三步所筛选的新抗原列表进行优化,用于评估新抗原在肿瘤中的真实免疫原性。随后,本研究通过收集的免疫原性的体内体外金标准数据集,TIL浸润数据集,TCGA肿瘤数据集,免疫检查点抑制剂治疗数据集全面验证了计算框架的有效性,并初步证明了本工作定义的新抗原免疫原性打分函数可以作为有效的肿瘤免疫治疗预后标志物,并且优于已知的TMB, TNB(Tumor neoantigen burden)等预后标志物。pTunes整个架构采用多线程并行加速,提高了高通量测序数据处理的速度,并提供docker版本供用户快速部署使用。
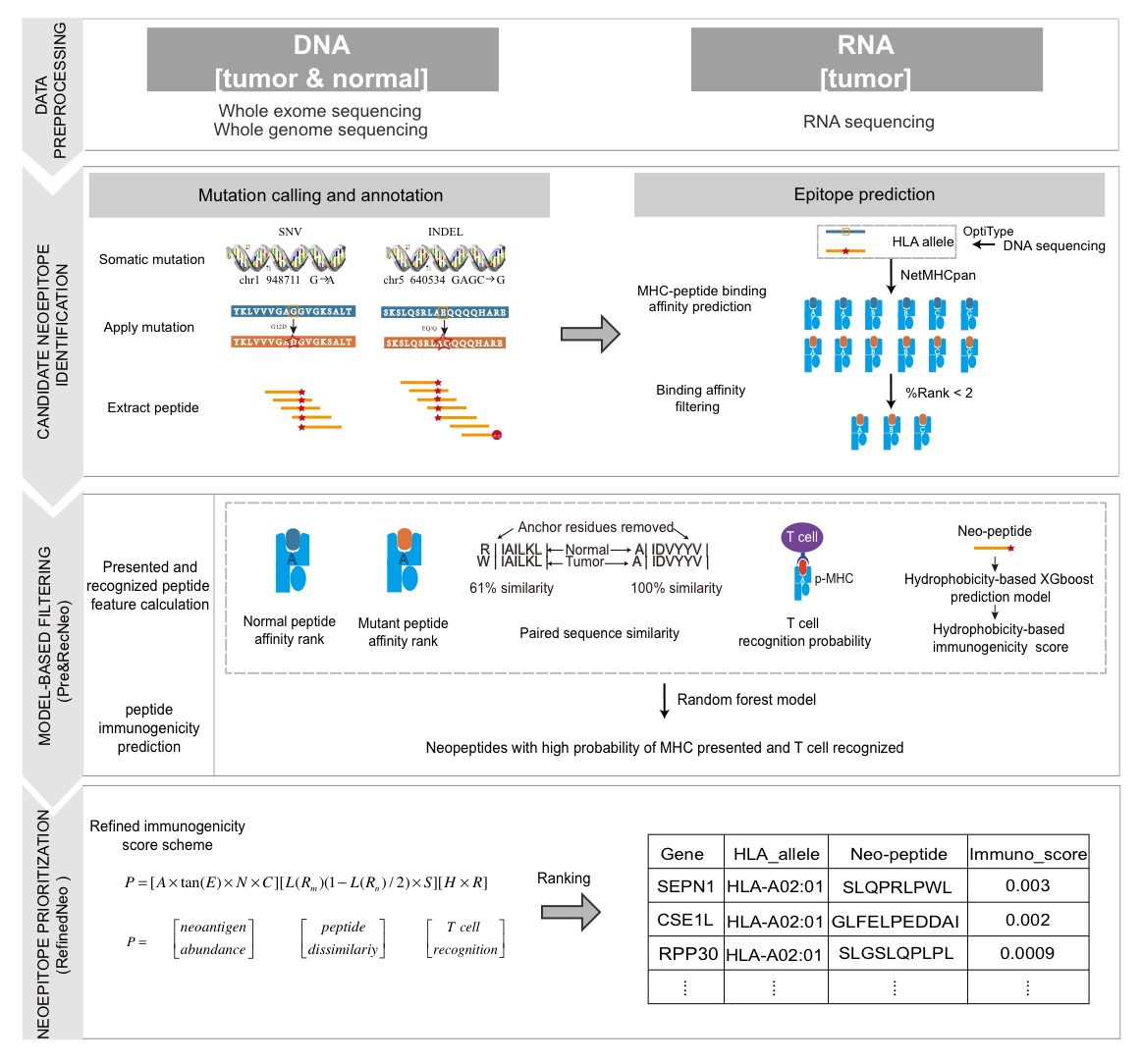
图一:pTuneos 计算流程
刘琦教授课题组以人工智能和组学分析计算技术为基础,重点关注于肿瘤精准医学,基因编辑和药物研发领域的交叉问题研究。目前针对肿瘤新抗原的计算筛选开展了一系列研究:包括系统研究基因融合(gene fusion)来源的新抗原在不同肿瘤中的分布情况及免疫原性【iScience,2019】【2】;系统探讨了不同来源肿瘤新抗原的计算及应用问题【Trends in Molecular Medicine,2019】【3】等。本工作第一作者是刘琦教授课题组博士周驰,通讯作者是同济大学刘琦教授。本研究成果得到了国家科技部重点研发计划精准医学重大专项,慢病专项,国家自然科学基金和上海市科委的基金项目资助。上海交通大学附属第九人民医院孙树洋教授课题组,上海生物信息中心谢鹭教授课题组及同济大学附属东方医院肿瘤科高勇主任等提供了有益的指导和帮助。
参考文献:
1. Zhou, C., Wei, Z., Zhang, Z. et al., Liu Q*, pTuneos: prioritizing tumor neoantigens from next-generation sequencing data. Genome Med 11, 67 (2019) doi:10.1186/s13073-019-0679-x.
2. Wei Z, Zhou C, Zhang Z, Guan M, Zhang C, Liu Z, Liu Q*, The landscape of tumor fusion neoantigens: a pan-cancer analysis. iScience, 2019.
3. Zhou C, Zhu C, Liu Q*, Toward in-silico identification of tumor neoantigens in immunotherapy. Trends in Molecular Medicine, 2019.
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


