同济大学曹志伟教授课题组研发糖蛋白抗原空间表位预测平台
外源性抗原入侵生物体时,抗原抗体的特异性结合是清除病原体的关键步骤,准确识别与抗体结合的空间表位对于疫苗的合理设计、免疫诊断和抗体筛选具有重要指导意义。传统表位鉴定方法通常需要大量实验,甚至结晶结构,耗时耗力,如果采用计算方法即可快速准确定位表位。以往的空间表位预测算法都聚焦于裸蛋白抗原,然而抗原蛋白的糖基化对表位影响很大,迫切需要适用于糖蛋白抗原的空间表位预测算法。
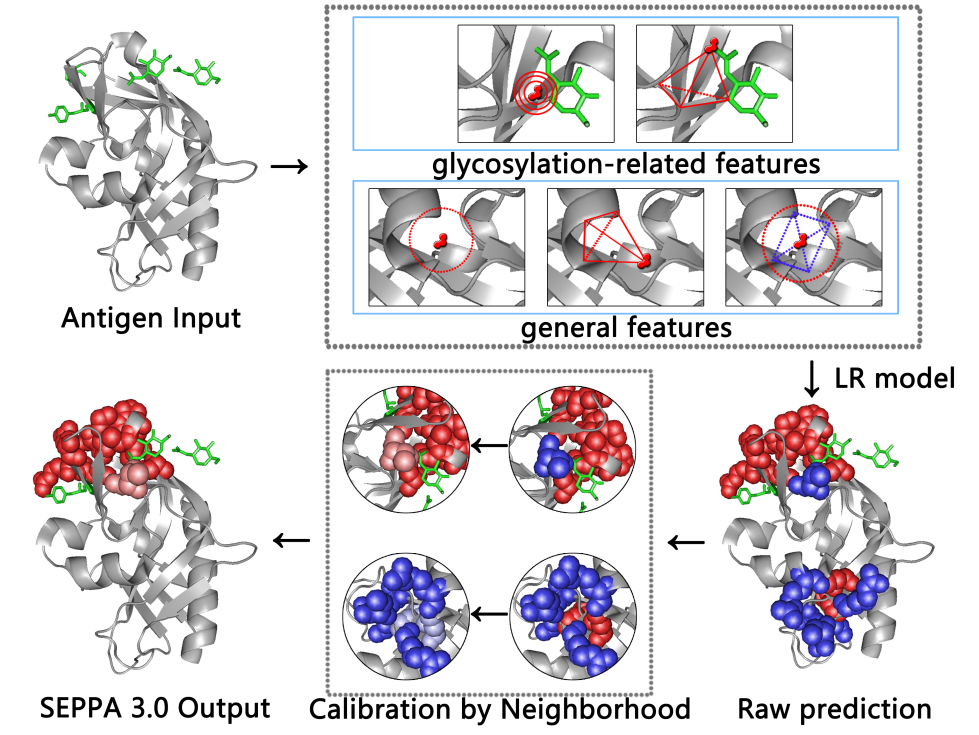
同济大学生命科学与技术学院曹志伟教授课题组长期致力于免疫识别的相关算法研究,曾于2009年和2014年在国际著名学术期刊《Nucleic Acids Research》上发表了分别表位预测在线工具SEPPA1.0【1】和SEPPA2.0【2】。SEPPA已在B细胞表位领域成为benchmark技术,近日该课题组在表位预测算法相关领域又取得新的突破。2019年5月22日,于《Nucleic Acids Research》上在线发表了题为”SEPPA 3.0—enhanced spatial epitope prediction enabling glycoprotein antigens”的研究论文。该工作中,研究人员充分考虑糖基化位点对空间微环境的影响,设计了糖基化相关的全新分类参数,同时引入反向“校正”,开发了糖蛋白抗原空间表位预测算法SEPPA 3.0。SEPPA 3.0计算平台的推出,不仅刷新了普通蛋白抗原空间表位预测的最高性能,还填补了糖蛋白抗原空间表位预测领域的空白,可为疫苗合理设计提供计算支持。
该工作在曹志伟教授与裘天颐博士共同指导下完成,曹志伟教授为主要通讯作者,裘天颐博士为共同通讯作者。曹志伟教授课题组的2016级硕士生周晨和2017级硕士生陈子鲲为本文的共同第一作者,同济大学为第一通讯单位。该研究得到了国家重点研发计划、中央高校交叉项目和上海市扬帆计划的共同支持。
【1】Sun J, et al, Cao ZW#. SEPPA: a computational server for spatial epitope prediction of protein antigens. Nucleic Acids Research, 37, W612-W616.
【2】Qi T, et al, Cao ZW#. SEPPA 2.0—more refined server to predict spatial epitope considering species of immune host and subcellular localization of protein antigen. Nucleic Acids Research, 42, W59-W63.
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


