科研进展
当Deep learning遇见CRISPR——同济大学课题组开发人工智能平台DeepCRISPR进行基因编辑...
发布时间:2018-06-25
CRISPR基因编辑与深度学习分别是当前生命科学和信息科学领域的热点研究技术。实现CRISPR基因敲除的重要挑战之一是设计具有高打靶活性的sgRNA,同时降低其全基因组范围内的脱靶,从而完成高效及特异性的基因敲除。近日,同济大学刘琦教授课题组及其合作者首次开发了一种基于人工智能深度学习框架的向导RNA(sgRNA)设计的计算平台DeepCRISPR。该平台基于深度学习模型进行一站式的sgRNA打靶活性预测及全基因组范围类的脱靶谱(off-target profile)预测,从而帮助用户挑选最优化的sgRNA进行基因编辑。相关成果发表在国际著名期刊《Genome Biology》。
DeepCRISPR平台基于深度学习模型,首次提出CRISPR系统的打靶活性预测和脱靶谱预测的统一计算框架(图一),并且在以下四个层面整合了若干有效的计算技术进行sgRNA的优化设计:
(1) DeepCRISPR平台整合了来自多细胞系的打靶活性数据及多个脱靶检测技术(GUIDE-seq、Digenome-seq、BLESS、HTGTS、IDLV)产生的脱靶数据。其整体架构基于卷积神经网络(CNN)进行模型训练,同时考虑了sgRNA序列层面和表观遗传层面(如染色质开放程度,甲基化等)的特征描述,利用深度学习layer-wise的表征学习(Representation Learning)能力自动学习有效的特征表示,避免人工进行sgRNA的特征工程,并且可以达到细胞系特异性的sgRNA设计。
(2) DeepCRISPR平台首次采用多细胞系genome-wide的非标记sgRNA序列(包含表观遗传层面信息,共~6.8亿样本)进行自编码式(Auto-encoder)的模型预训练(Pre-training)。该预训练模型从海量的无标记样本中进行sgRNA的特征学习,一定程度上避免了在有限标记样本下的深度学习过拟合问题。
(3) DeepCRISPR平台首次借鉴了图像处理领域的样本扩增技术(Data Augmentation)进行标记sgRNA样本的扩增(例如改变sgRNA远离PAM端的碱基并不改变该sgRNA的活性,类比于图像处理中改变图像中若干像素并不改变该图像的标签),从而有效的扩展了标记训练样本的数量。
(4) DeepCRISPR平台将Bootstrap重采样技术应用至深度学习的mini-batch过程中,缓解了脱靶预测中的数据不平衡性(Data Imbalance),提升了脱靶预测的精度。
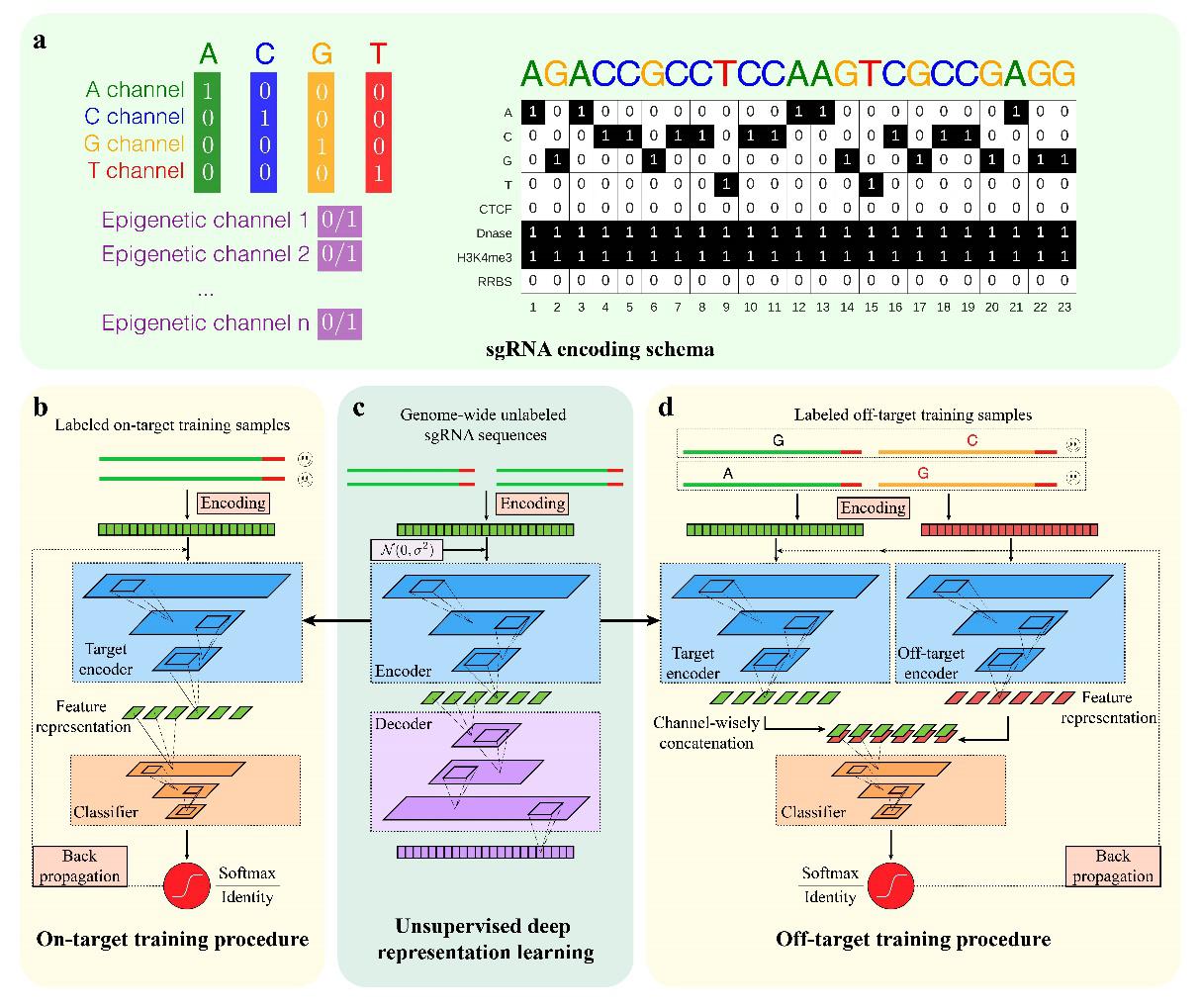
图一:DeepCRISPR计算框架
通过面向基准数据的系统测试,DeepCRISPR超越了现有代表性的打靶活性预测及全基因组脱靶预测工具(如sgRNA designer、CFD score,Nature Biotechnology 2016)。DeepCRISPR底层基于Tensorflow开发,目前提供网页版(图二)和Github版本,同时可对sgRNA的全基因组层面的脱靶预测结果进行可视化展示。

图二:DeepCRISPR网页版本
刘琦教授课题组以AI和机器学习计算技术为基础,重点关注于生物医药大数据挖掘领域的交叉问题研究。目前主要关注于基因编辑的小RNA设计、药物信息学及肿瘤的精准治疗和免疫治疗。课题组围绕CRISPR系统的打靶预测和脱靶分析进行了系列工作:包括系统研究了CRISPR基因敲除过程中microhomology和in-frame mutation发生率之间的关系及开发了相关计算工具CAGE【Molecular Therapy-Nucleic Acids, 2016】;合作开发了包含非编码区的sgRNA设计工具CRISPR-DO【Bioinformatics, 2016】;系统探讨了CRISPR基因编辑系统中的in-silico sgRNA设计问题【Trends in Biotechnology, 2016】;对于主流的sgRNA打靶预测工具进行了基准评估【Briefings in Bioinformatics, 2017】等。本工作由同济大学刘琦教授、电信学院黄徳双教授、阿斯利康制药公司及美国麻省大学医学院马涵慧博士等合作完成。第一作者为同济大学博士研究生啜国晖和马涵慧博士。本项目得到了国家科技部重点研发计划精准医学重大专项,慢病专项,国家自然科学基金和上海市科委的基金项目资助。
Copyright© 2011-2015 生命科学与技术学院, All rights reserved
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


